install.packages("rattle")
install.packages("MVN")
install.packages("heplots")Analisis Diskriminan dengan tidymodels
library(lares)
library(discrim)
library(tidyverse)
library(tidymodels)
library(themis)
library(tidyposterior)
library(SmartEDA)
library(DataExplorer)
library(skimr)
library(ggpubr)
library(workflowsets)Deskripsi singkat data
Dataset wine berisi hasil analisis kimiawi dari anggur yang ditanam di daerah tertentu di Italia. Tiga jenis anggur diwakili dalam 178 sampel, dengan hasil 13 analisis kimia yang dicatat untuk setiap sampel. Variabel Type telah diubah menjadi variabel kategorik.
data ini bisa diperoleh dari package rattle
Import data di R
data(wine, package='rattle')
df <- wine
rm(wine)
glimpse(df)Rows: 178
Columns: 14
$ Type <fct> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, …
$ Alcohol <dbl> 14.23, 13.20, 13.16, 14.37, 13.24, 14.20, 14.39, 14.06…
$ Malic <dbl> 1.71, 1.78, 2.36, 1.95, 2.59, 1.76, 1.87, 2.15, 1.64, …
$ Ash <dbl> 2.43, 2.14, 2.67, 2.50, 2.87, 2.45, 2.45, 2.61, 2.17, …
$ Alcalinity <dbl> 15.6, 11.2, 18.6, 16.8, 21.0, 15.2, 14.6, 17.6, 14.0, …
$ Magnesium <int> 127, 100, 101, 113, 118, 112, 96, 121, 97, 98, 105, 95…
$ Phenols <dbl> 2.80, 2.65, 2.80, 3.85, 2.80, 3.27, 2.50, 2.60, 2.80, …
$ Flavanoids <dbl> 3.06, 2.76, 3.24, 3.49, 2.69, 3.39, 2.52, 2.51, 2.98, …
$ Nonflavanoids <dbl> 0.28, 0.26, 0.30, 0.24, 0.39, 0.34, 0.30, 0.31, 0.29, …
$ Proanthocyanins <dbl> 2.29, 1.28, 2.81, 2.18, 1.82, 1.97, 1.98, 1.25, 1.98, …
$ Color <dbl> 5.64, 4.38, 5.68, 7.80, 4.32, 6.75, 5.25, 5.05, 5.20, …
$ Hue <dbl> 1.04, 1.05, 1.03, 0.86, 1.04, 1.05, 1.02, 1.06, 1.08, …
$ Dilution <dbl> 3.92, 3.40, 3.17, 3.45, 2.93, 2.85, 3.58, 3.58, 2.85, …
$ Proline <int> 1065, 1050, 1185, 1480, 735, 1450, 1290, 1295, 1045, 1…Eksplorasi Data
df %>%
skim_without_charts()| Name | Piped data |
| Number of rows | 178 |
| Number of columns | 14 |
| _______________________ | |
| Column type frequency: | |
| factor | 1 |
| numeric | 13 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| Type | 0 | 1 | FALSE | 3 | 2: 71, 1: 59, 3: 48 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 |
|---|---|---|---|---|---|---|---|---|---|
| Alcohol | 0 | 1 | 13.00 | 0.81 | 11.03 | 12.36 | 13.05 | 13.68 | 14.83 |
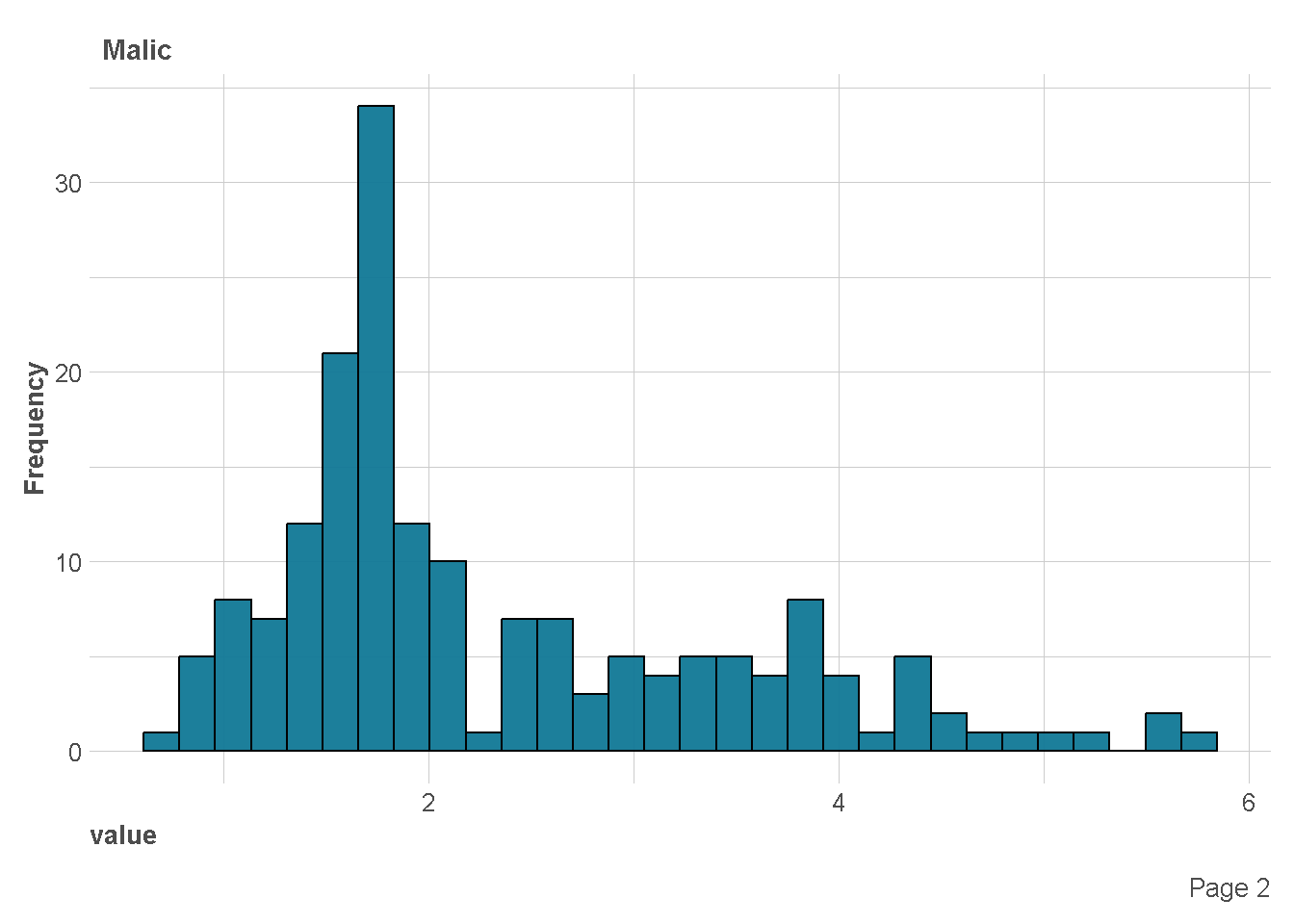
| Malic | 0 | 1 | 2.34 | 1.12 | 0.74 | 1.60 | 1.87 | 3.08 | 5.80 |
| Ash | 0 | 1 | 2.37 | 0.27 | 1.36 | 2.21 | 2.36 | 2.56 | 3.23 |
| Alcalinity | 0 | 1 | 19.49 | 3.34 | 10.60 | 17.20 | 19.50 | 21.50 | 30.00 |
| Magnesium | 0 | 1 | 99.74 | 14.28 | 70.00 | 88.00 | 98.00 | 107.00 | 162.00 |
| Phenols | 0 | 1 | 2.30 | 0.63 | 0.98 | 1.74 | 2.36 | 2.80 | 3.88 |
| Flavanoids | 0 | 1 | 2.03 | 1.00 | 0.34 | 1.20 | 2.13 | 2.88 | 5.08 |
| Nonflavanoids | 0 | 1 | 0.36 | 0.12 | 0.13 | 0.27 | 0.34 | 0.44 | 0.66 |
| Proanthocyanins | 0 | 1 | 1.59 | 0.57 | 0.41 | 1.25 | 1.56 | 1.95 | 3.58 |
| Color | 0 | 1 | 5.06 | 2.32 | 1.28 | 3.22 | 4.69 | 6.20 | 13.00 |
| Hue | 0 | 1 | 0.96 | 0.23 | 0.48 | 0.78 | 0.96 | 1.12 | 1.71 |
| Dilution | 0 | 1 | 2.61 | 0.71 | 1.27 | 1.94 | 2.78 | 3.17 | 4.00 |
| Proline | 0 | 1 | 746.89 | 314.91 | 278.00 | 500.50 | 673.50 | 985.00 | 1680.00 |
Distribusi Variabel Kategorik
df %>%
ExpCatViz(clim = 10,
col = "#107896",
Page = NULL,
Flip = TRUE)[[1]]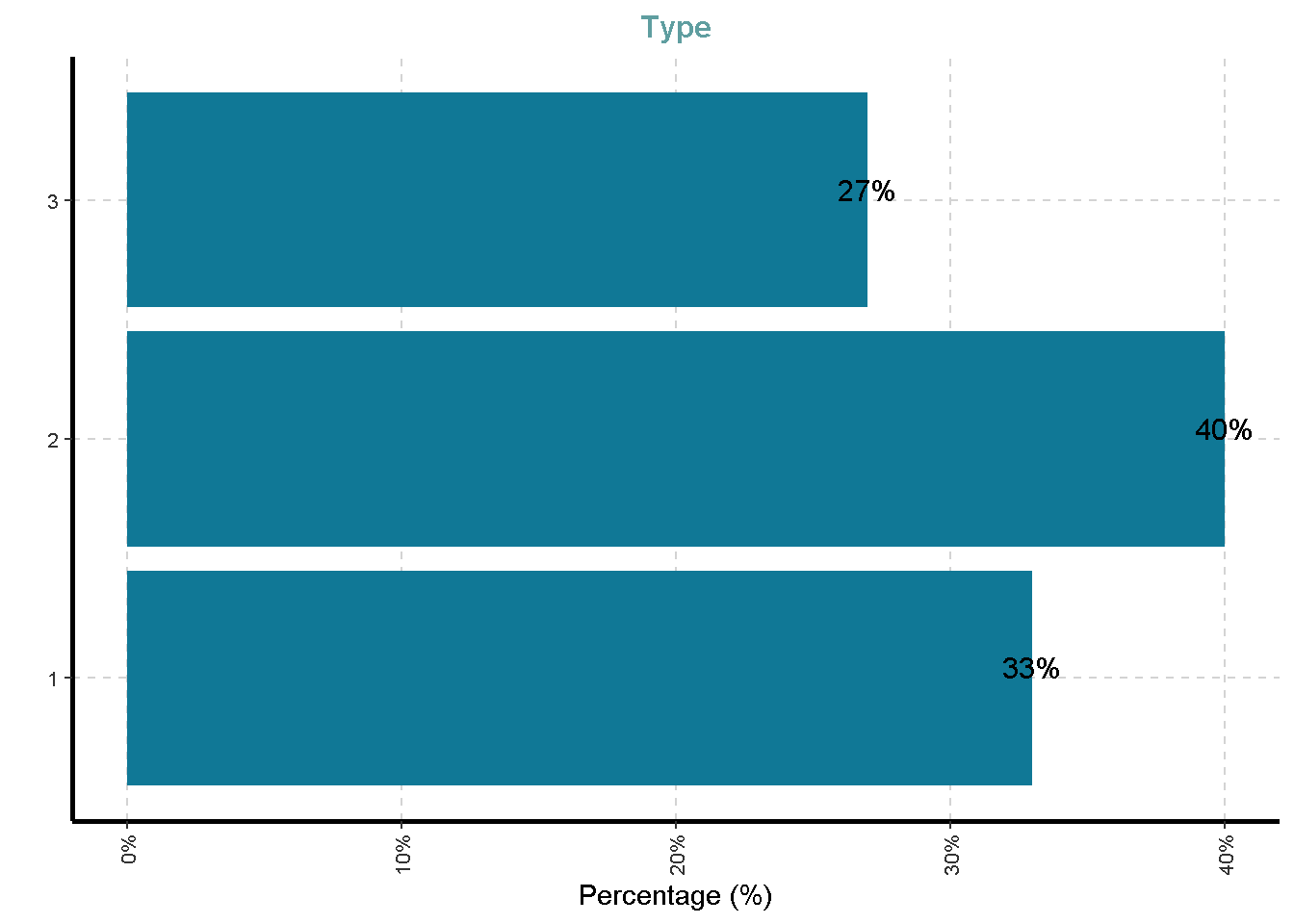
Distribusi Variabel Numerik
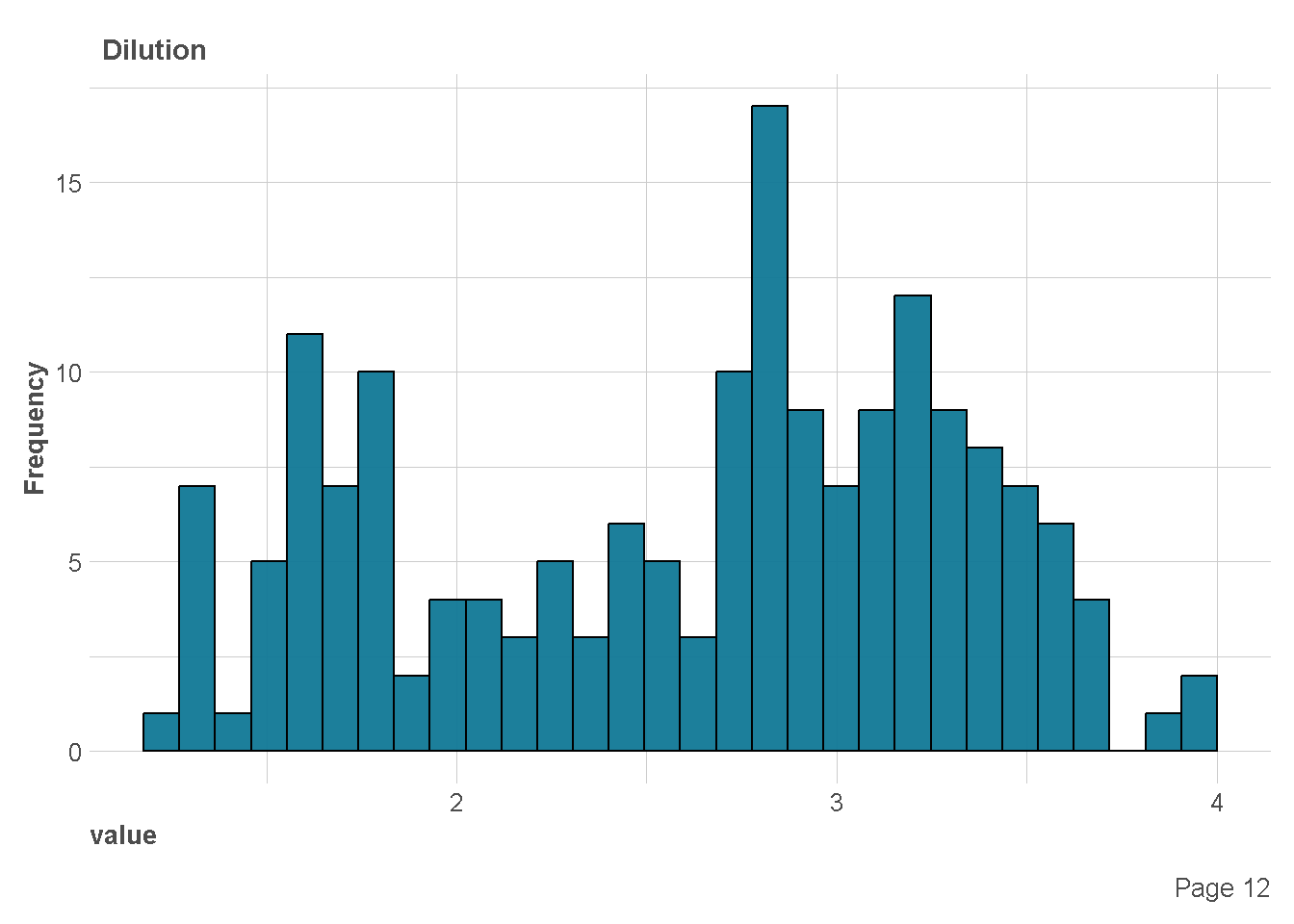
df %>%
plot_histogram(
ggtheme = theme_lares(),
geom_histogram_args = list(bins=30,
col="black",
fill="#107896"),
ncol = 1,nrow = 1)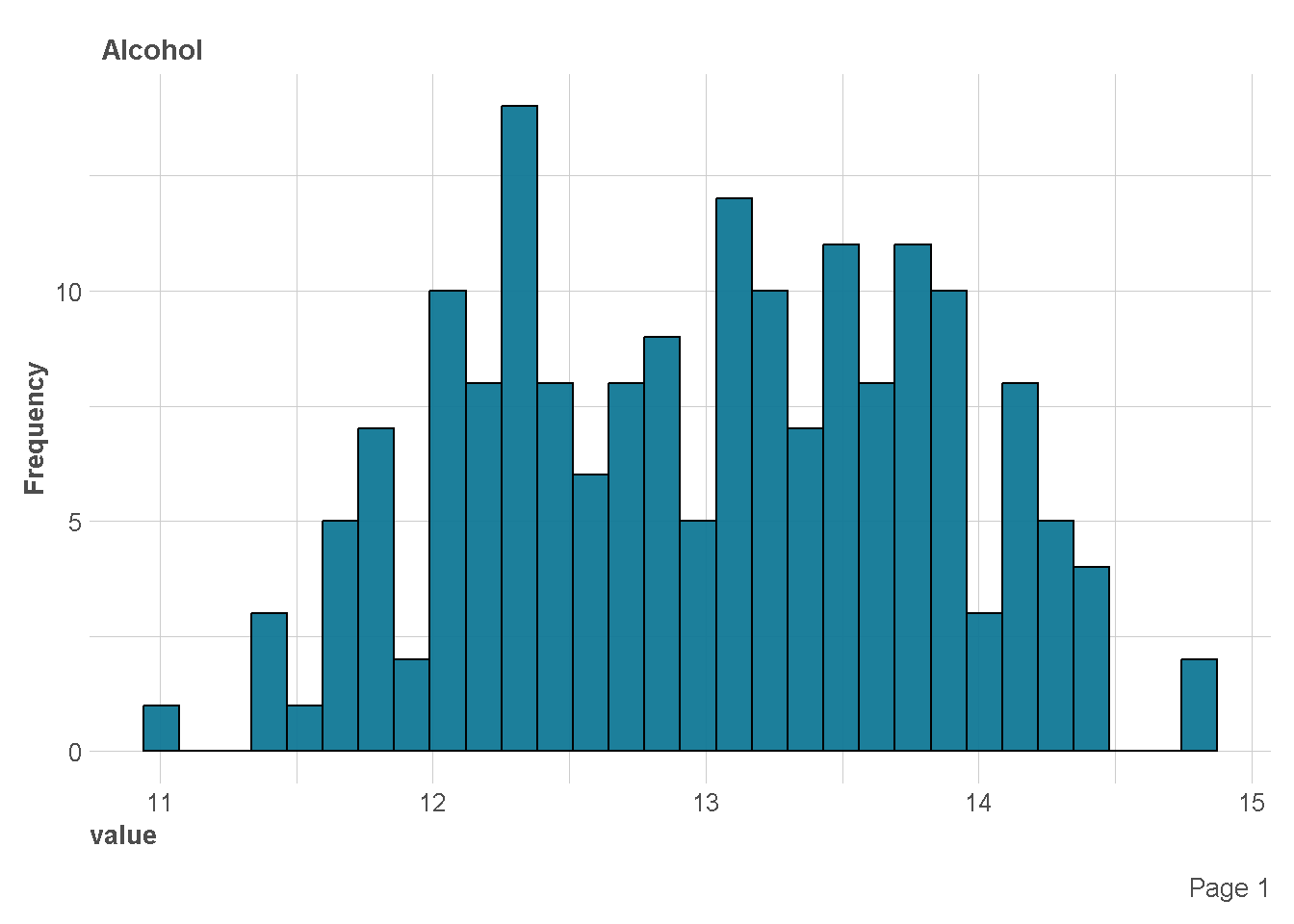
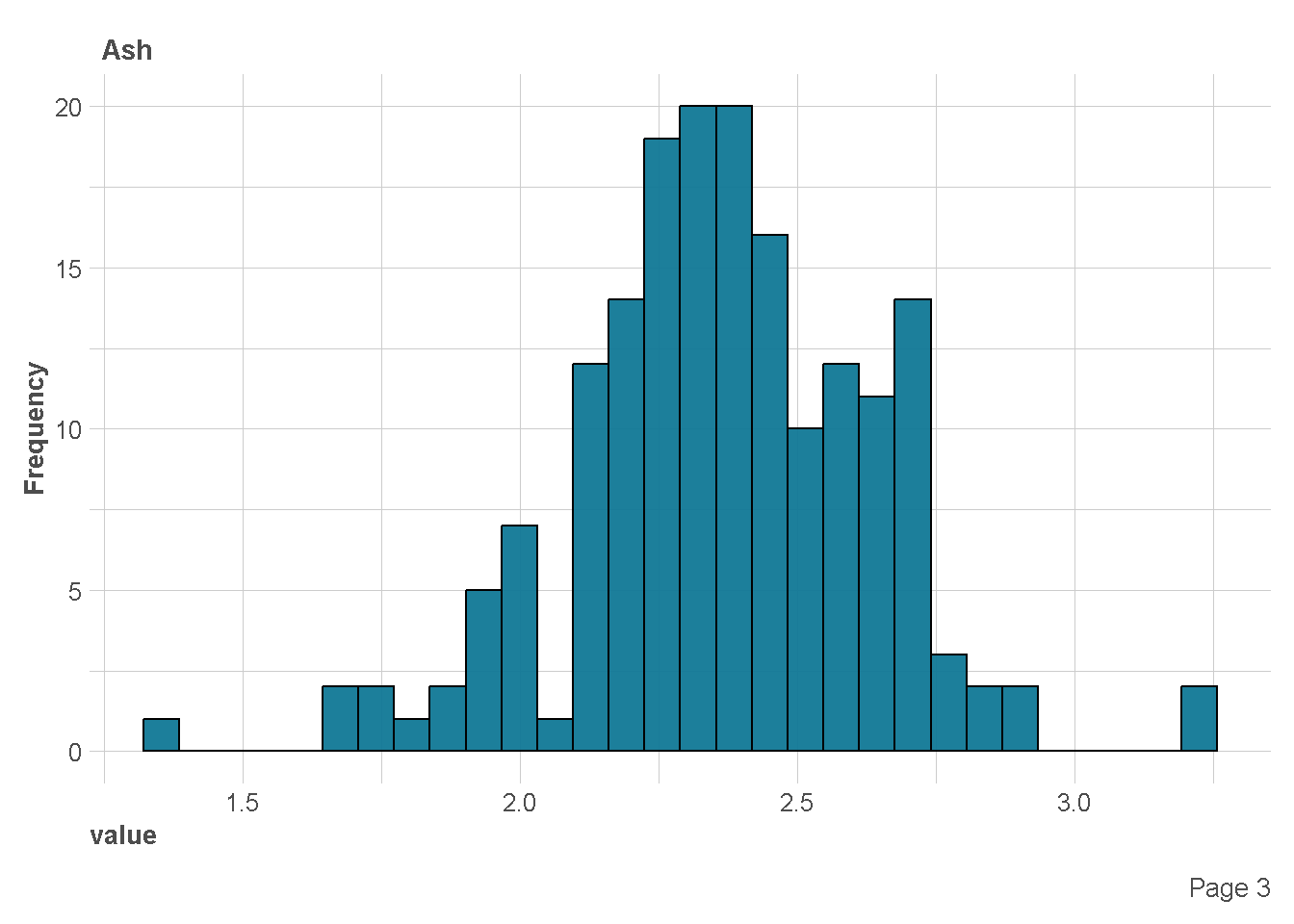
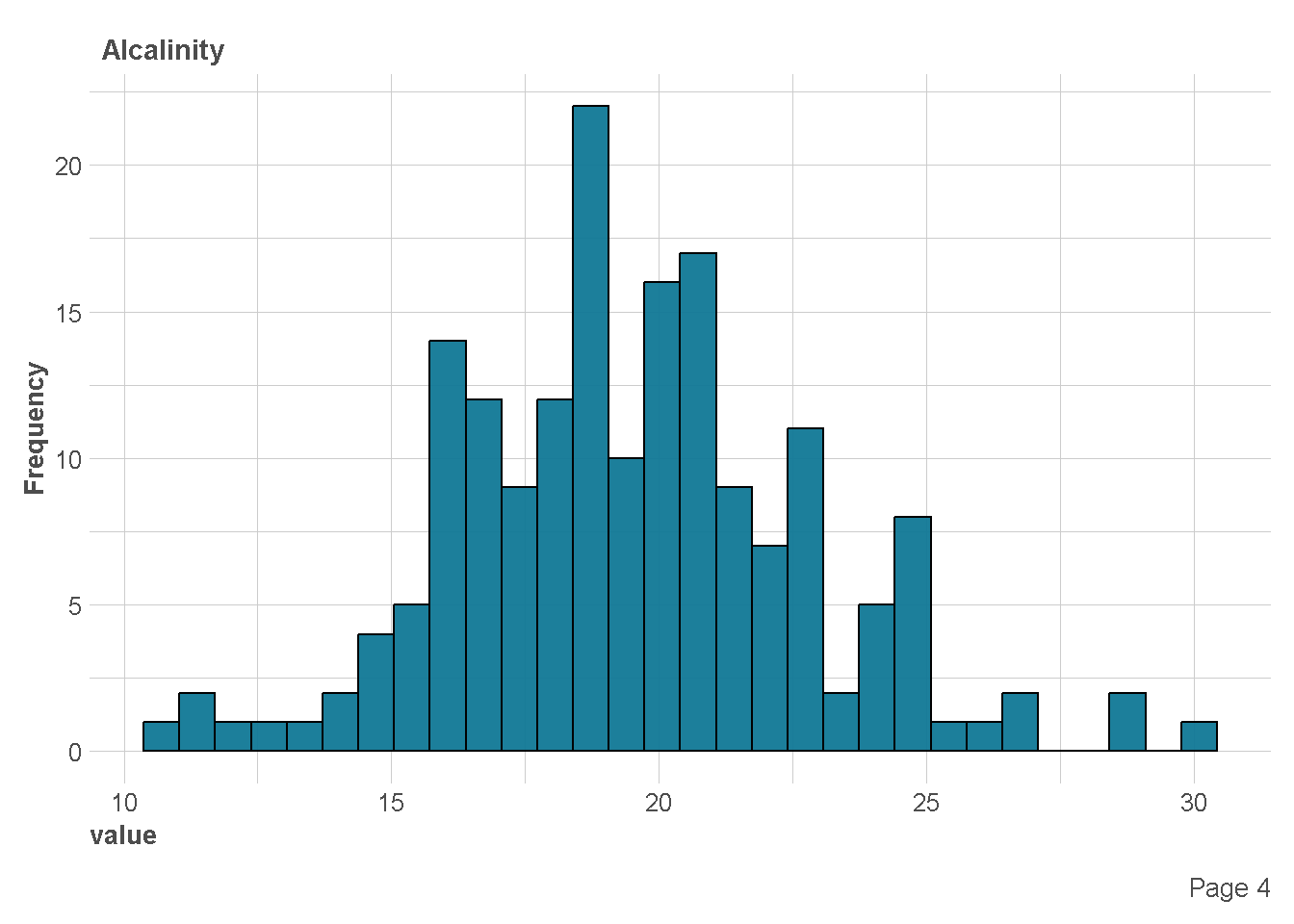
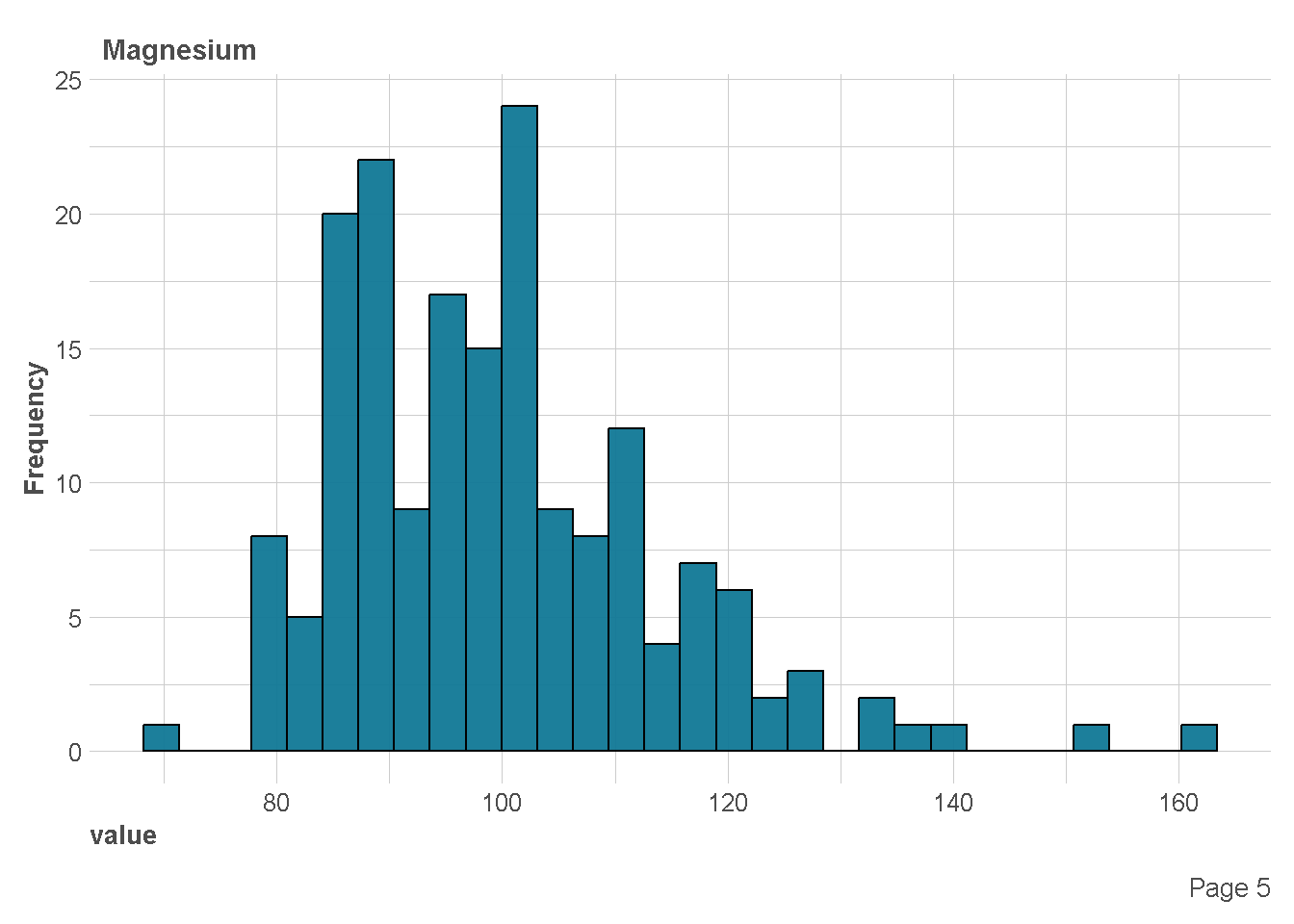
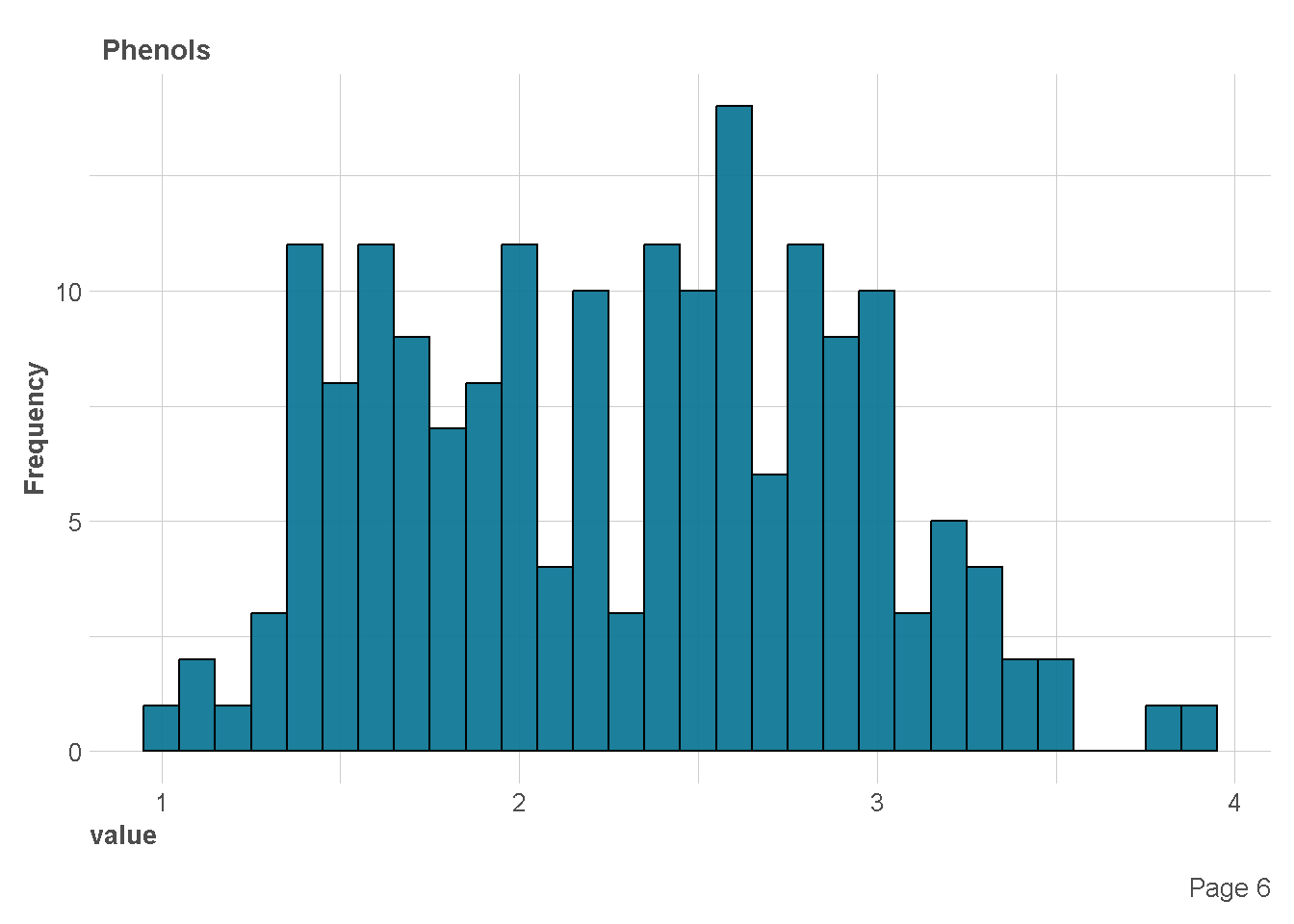
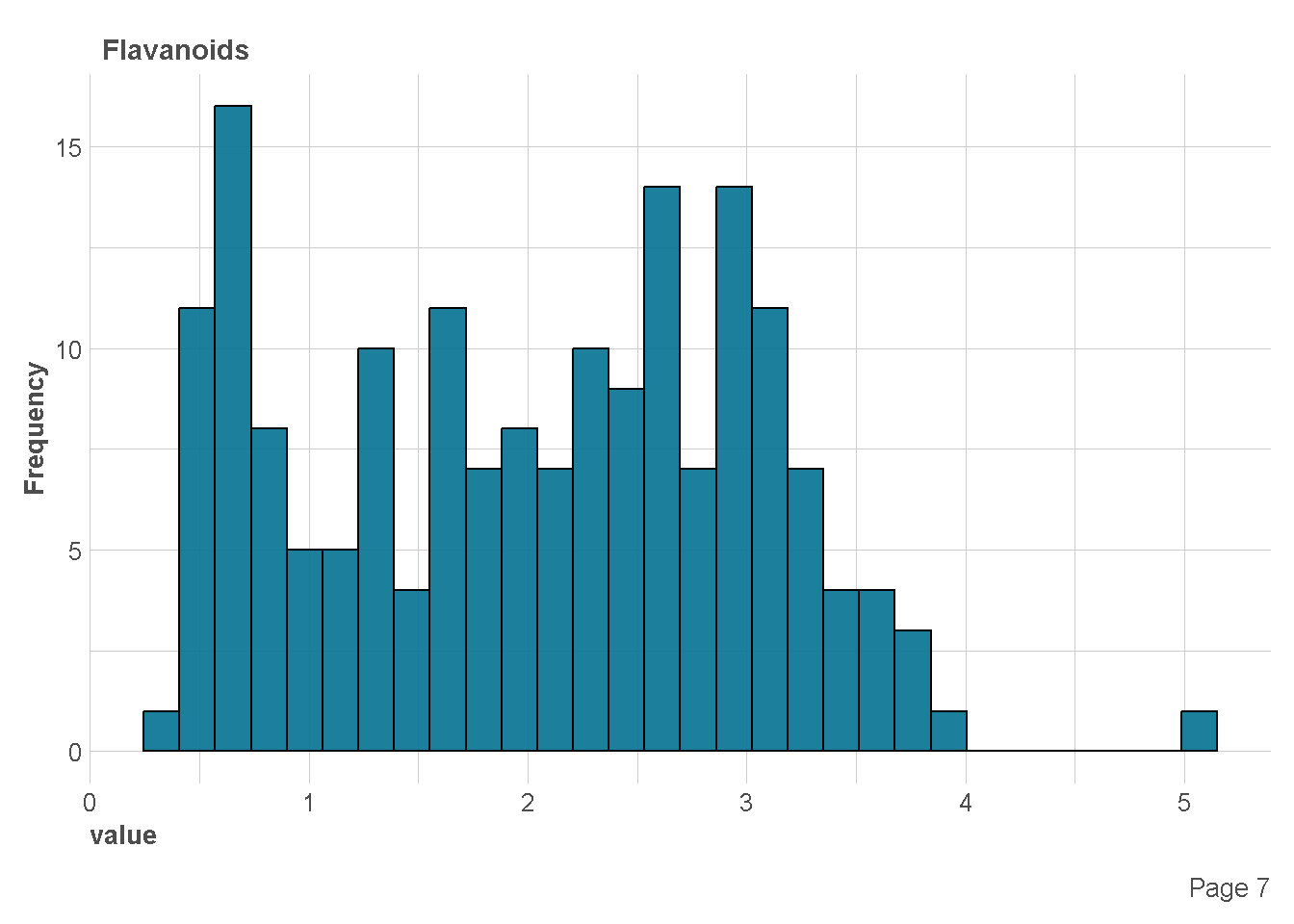
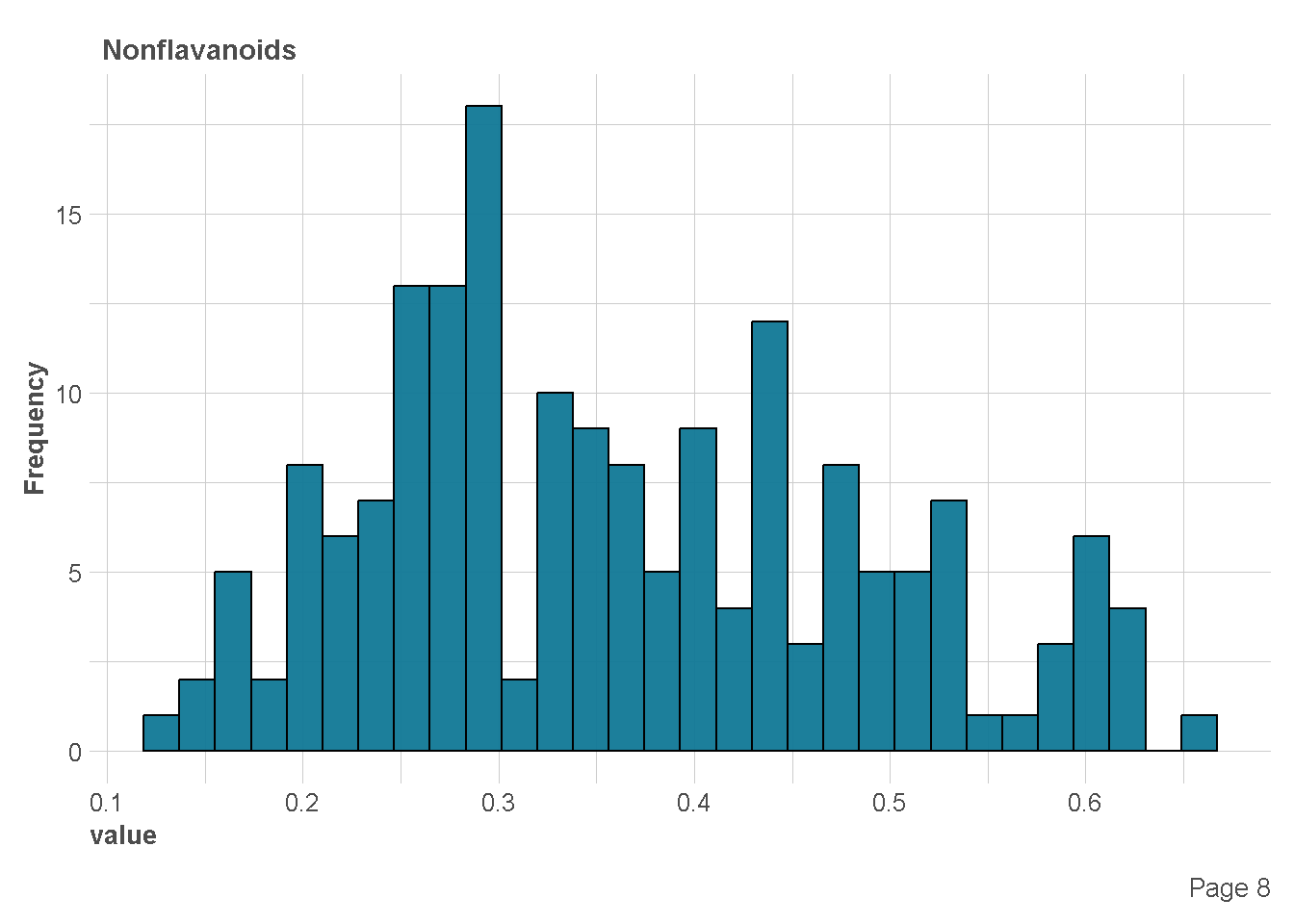
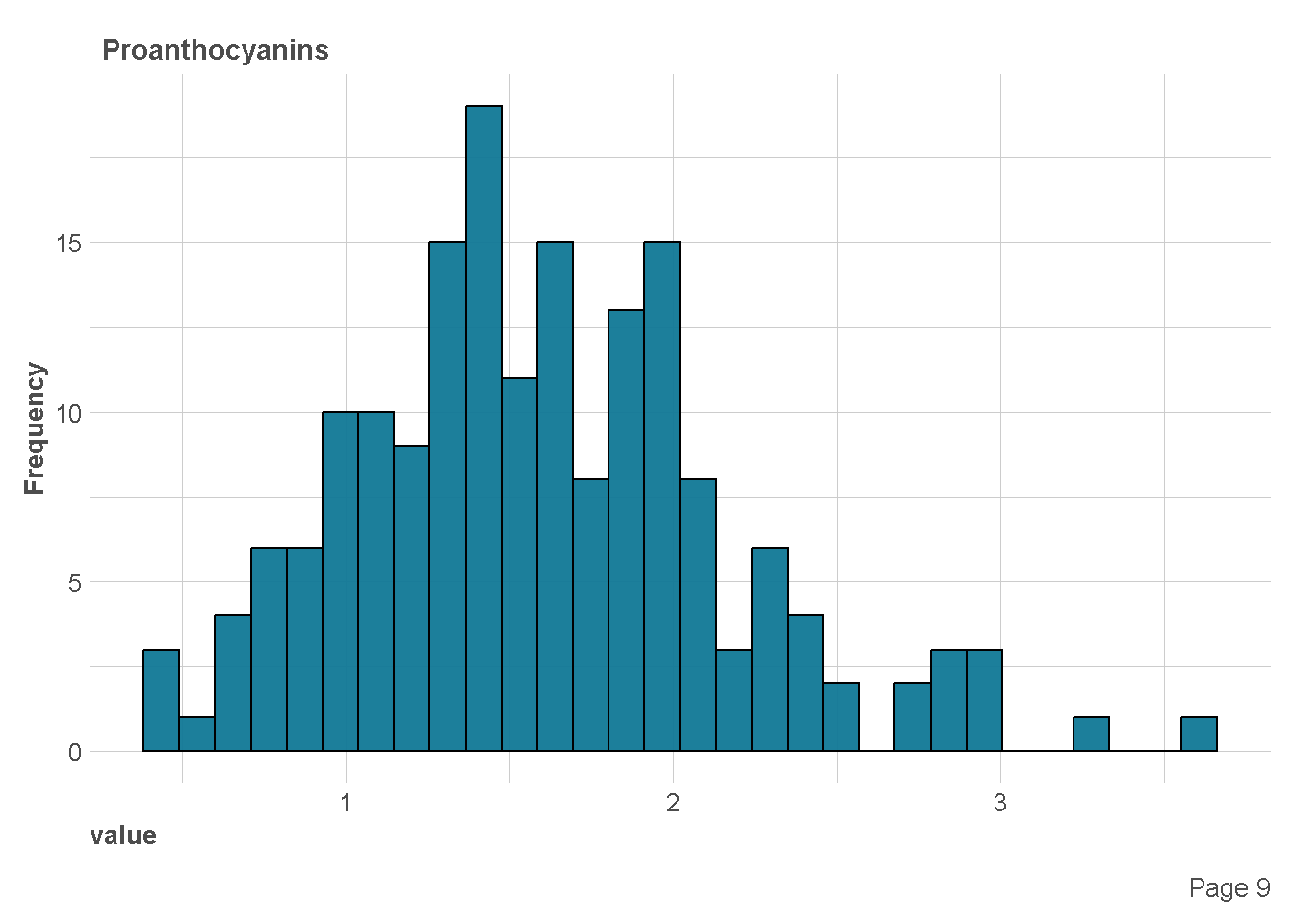
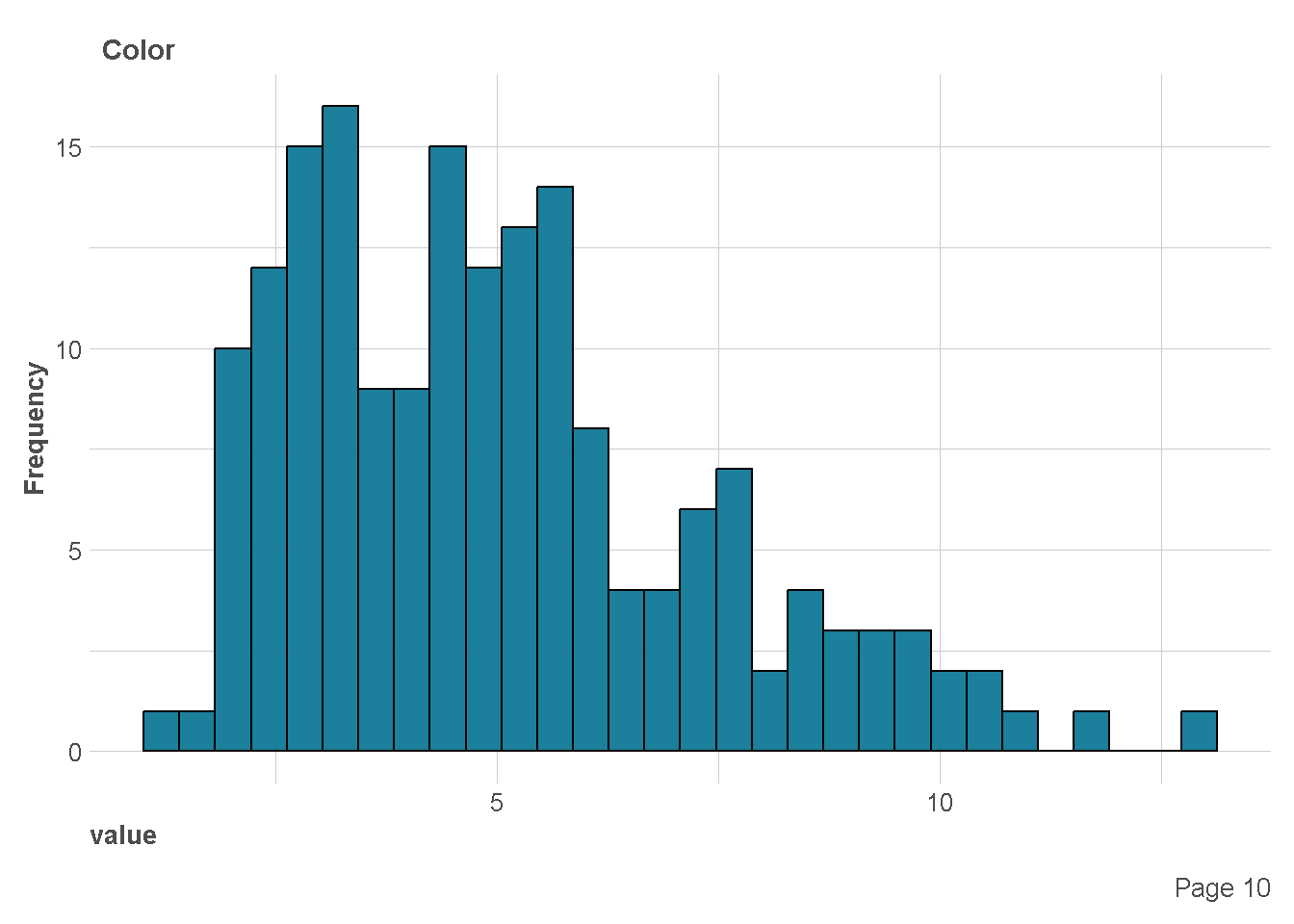
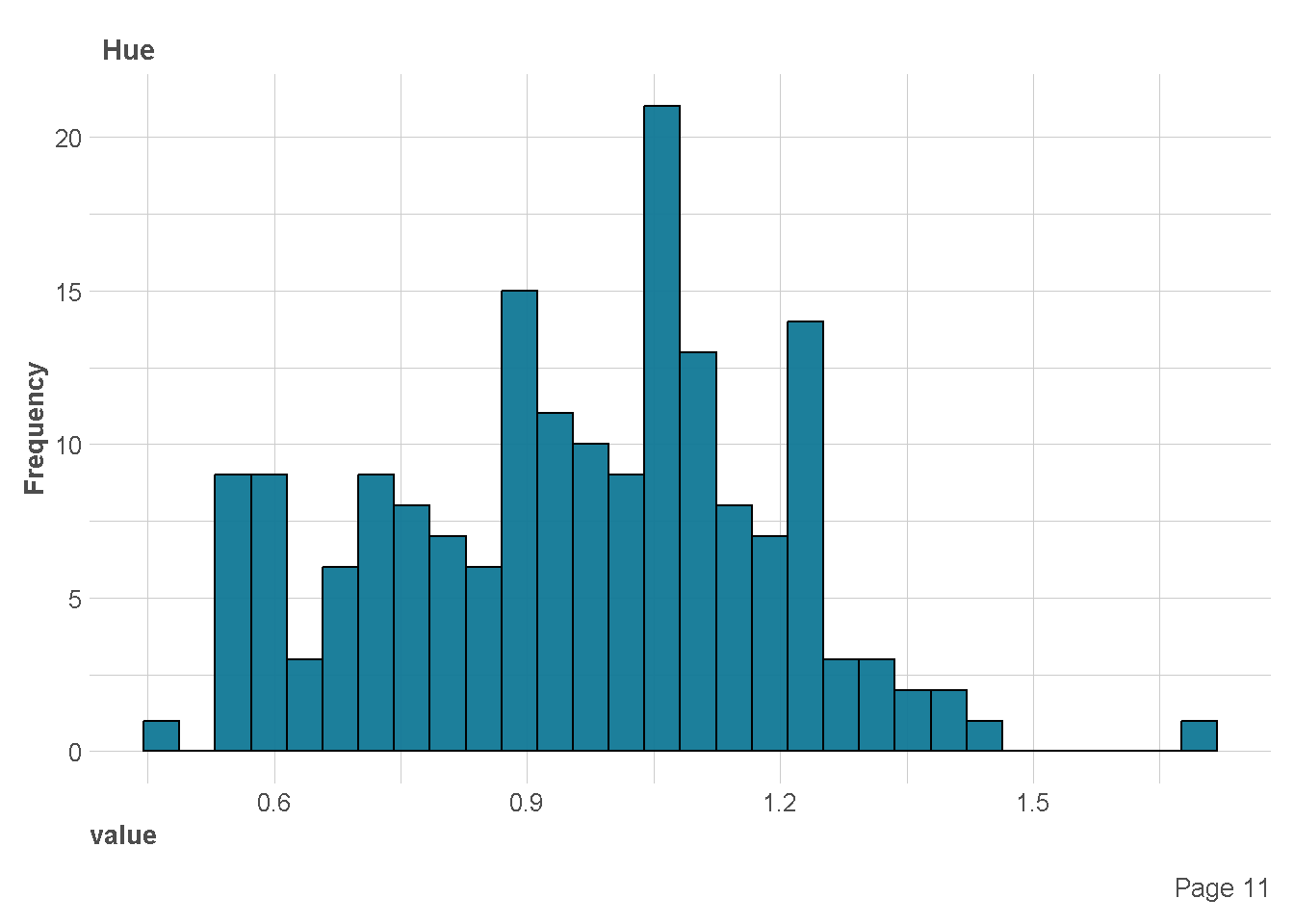

Ekplorasi Kategorik vs Numerik
df %>%
ExpNumViz(target = "Type",
type = 2)[[1]]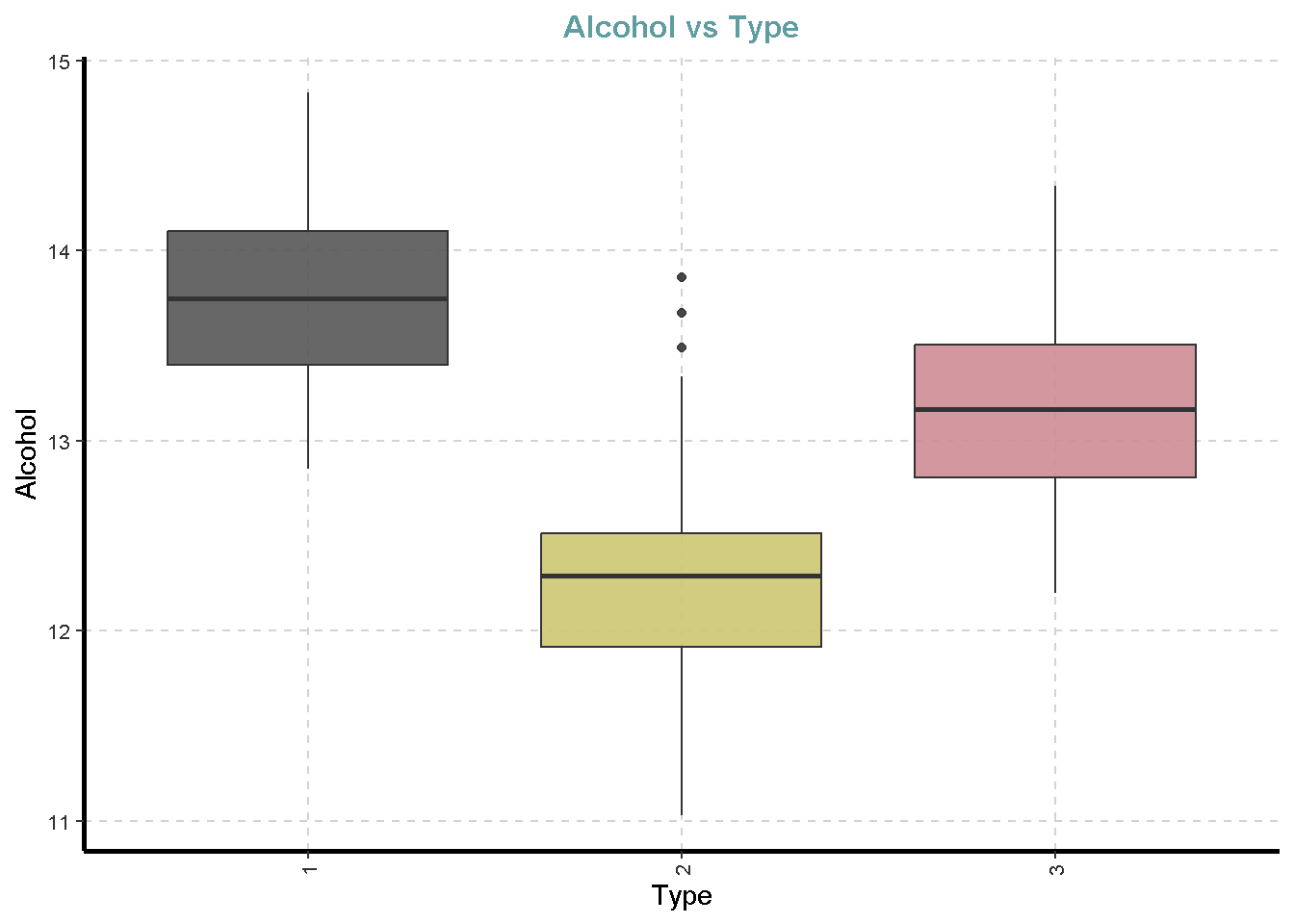
[[2]]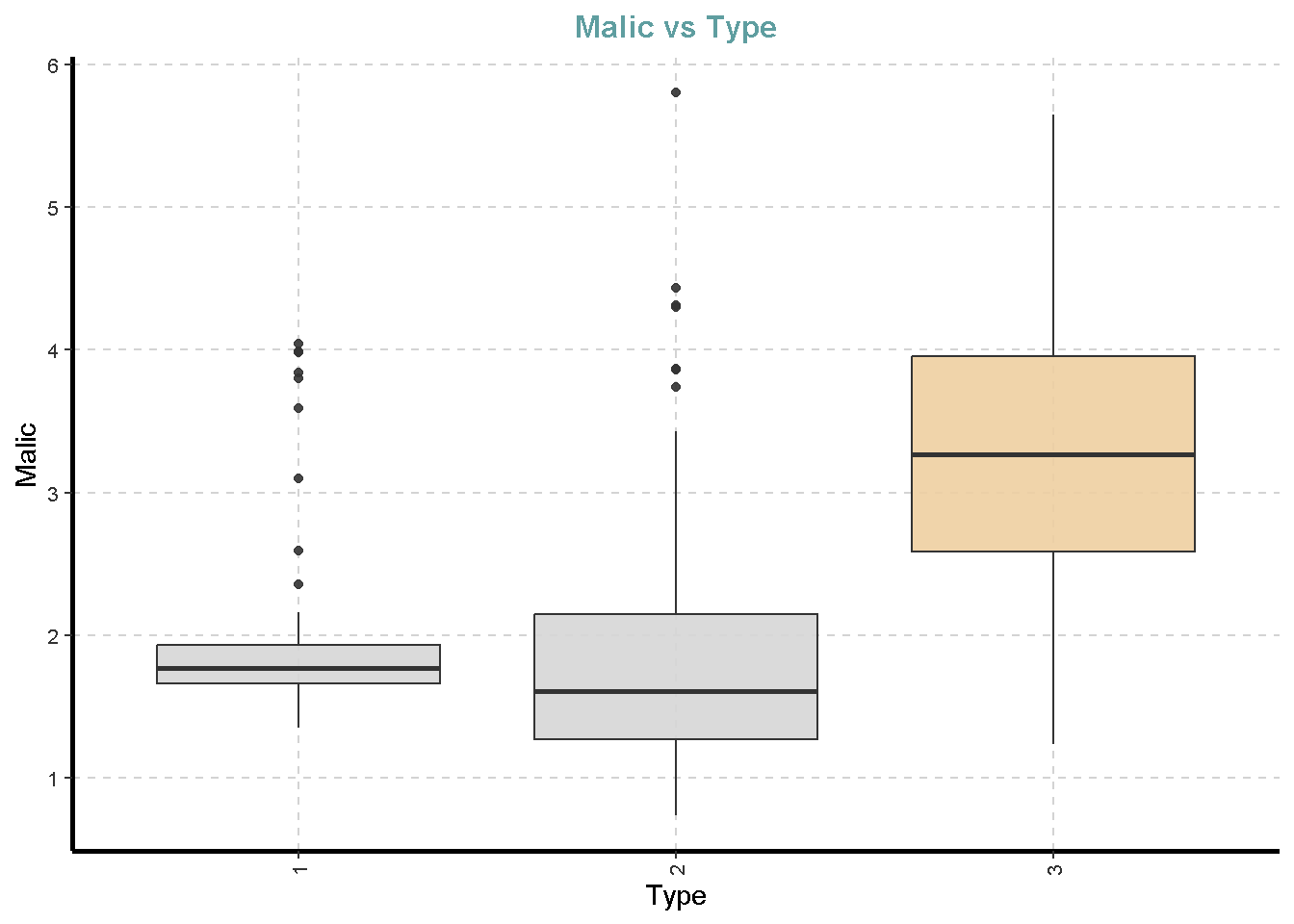
[[3]]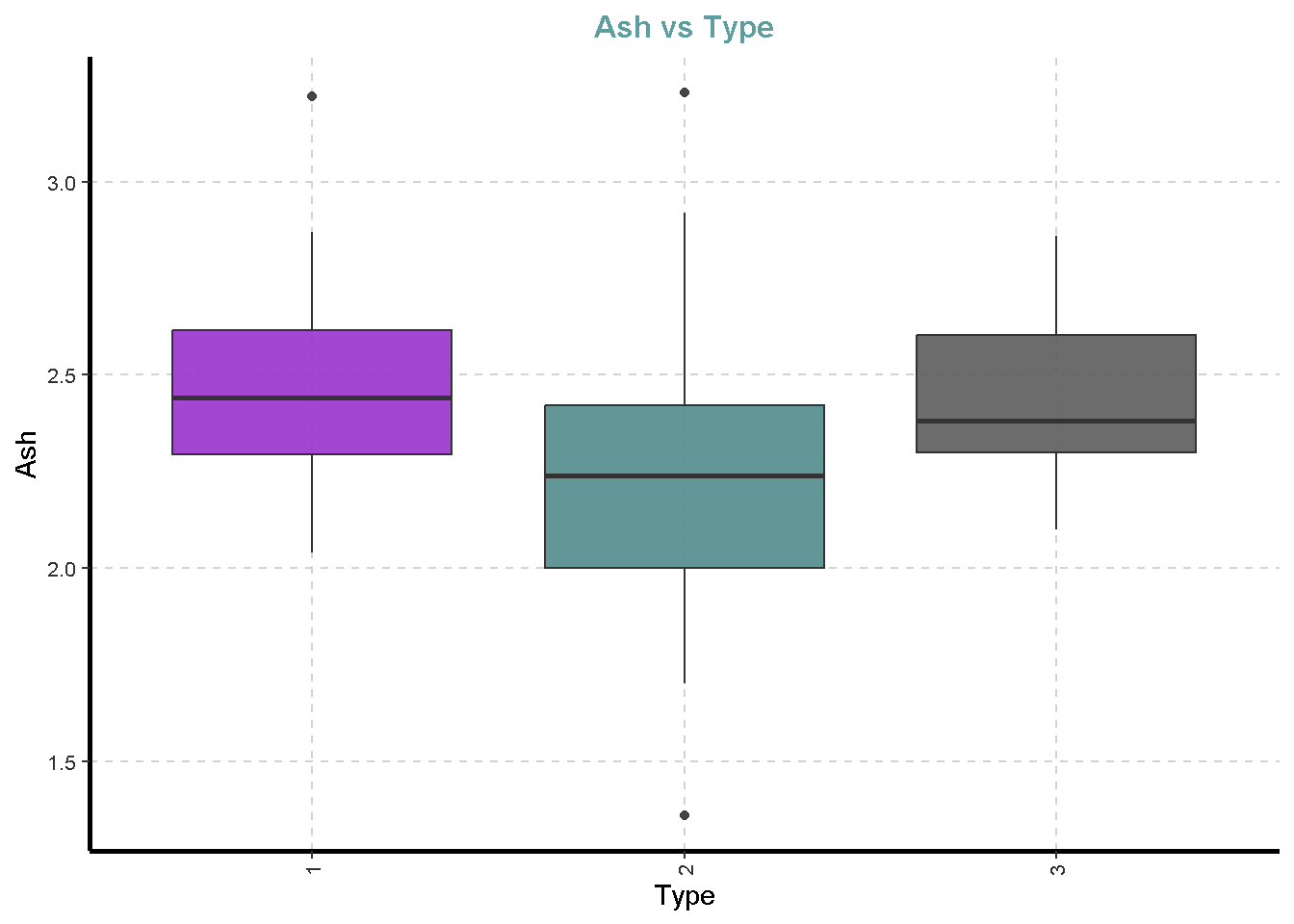
[[4]]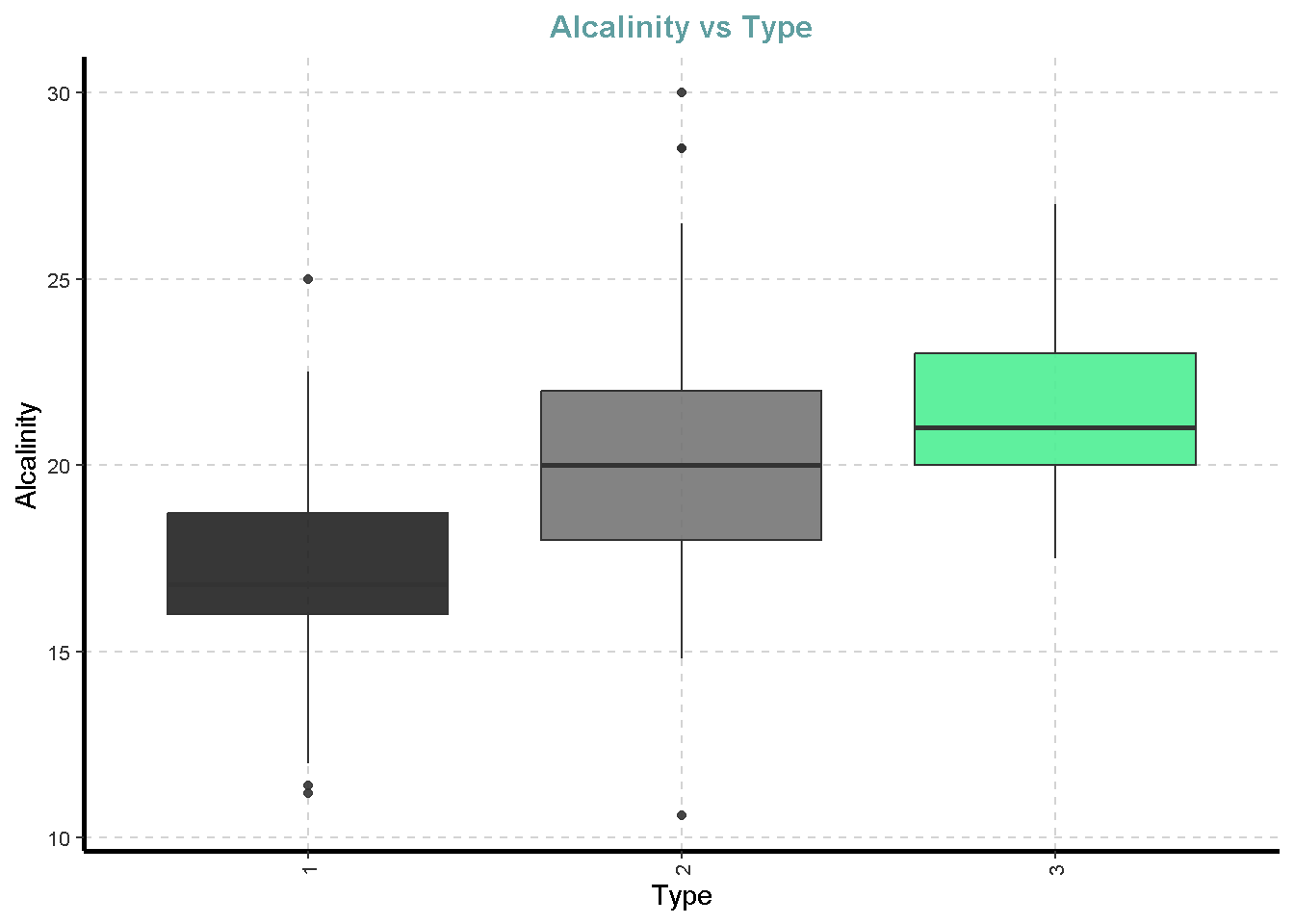
[[5]]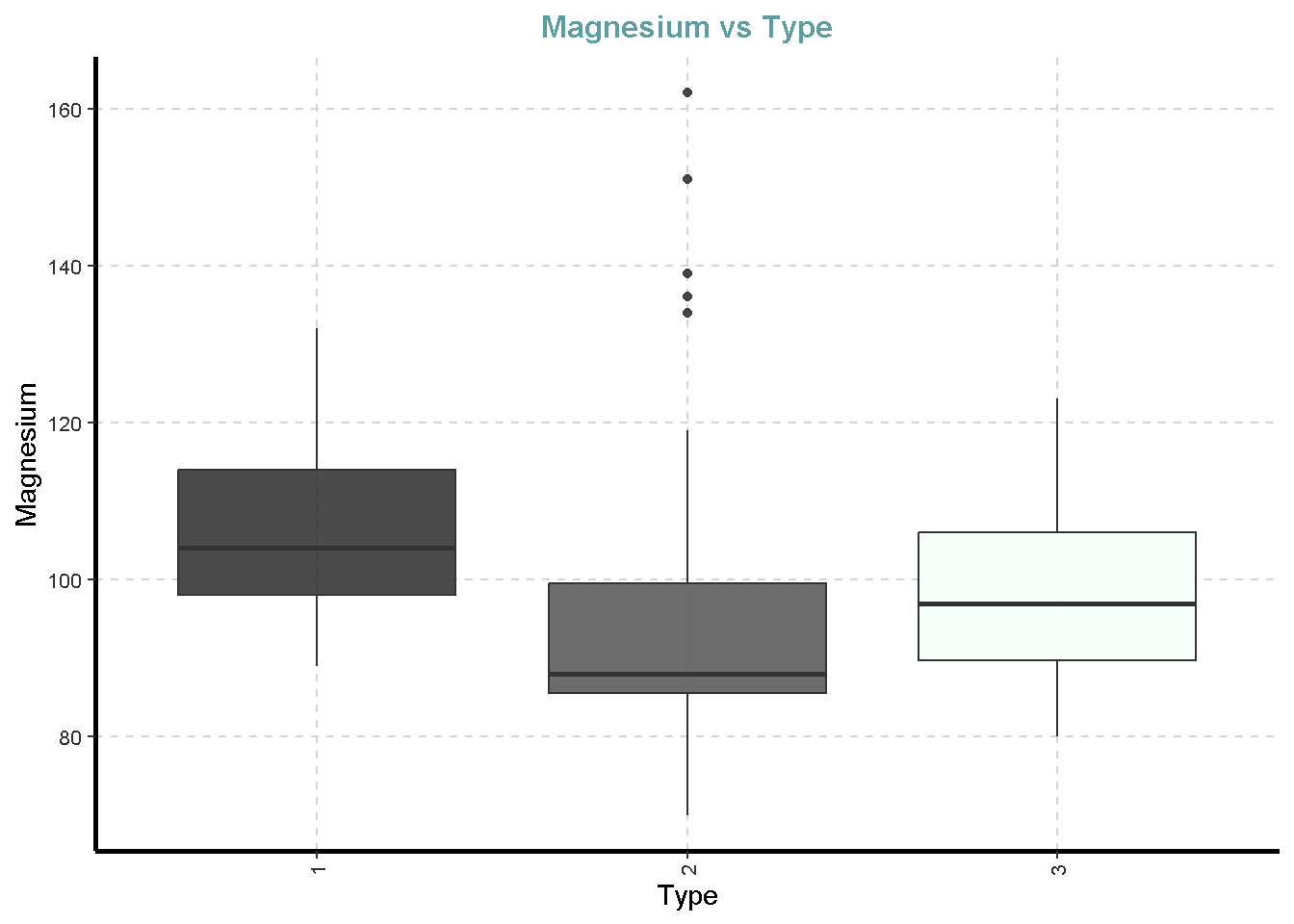
[[6]]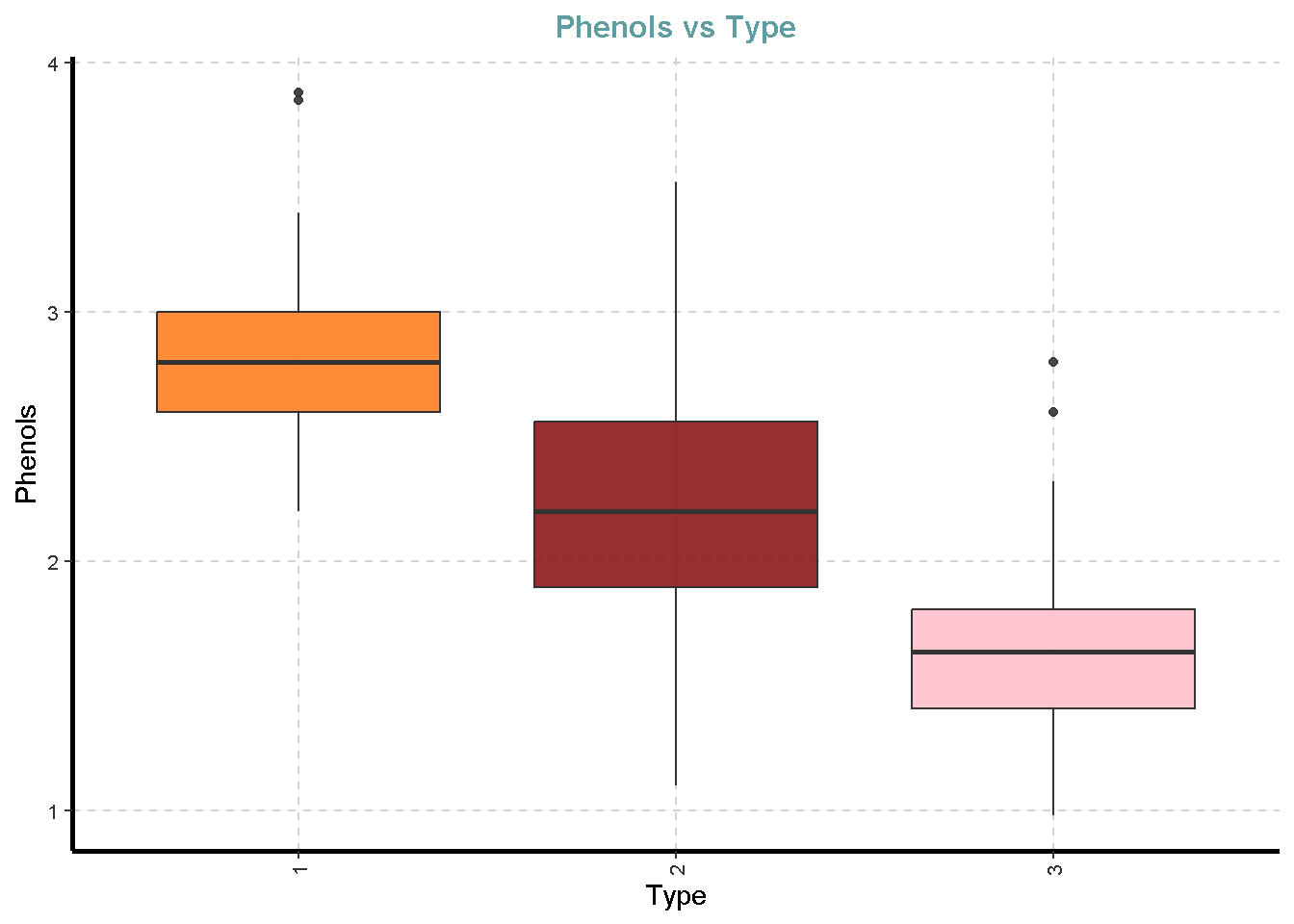
[[7]]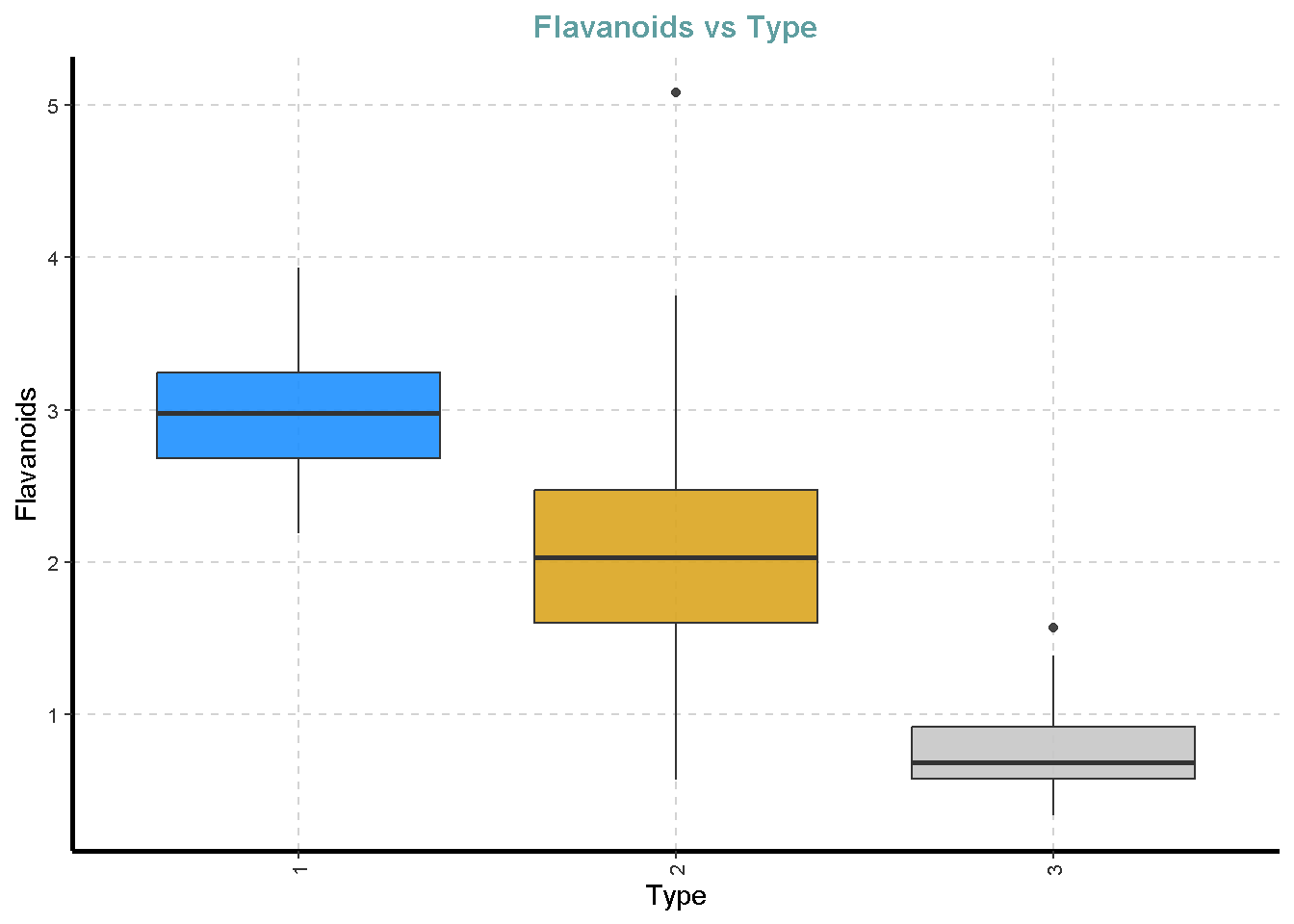
[[8]]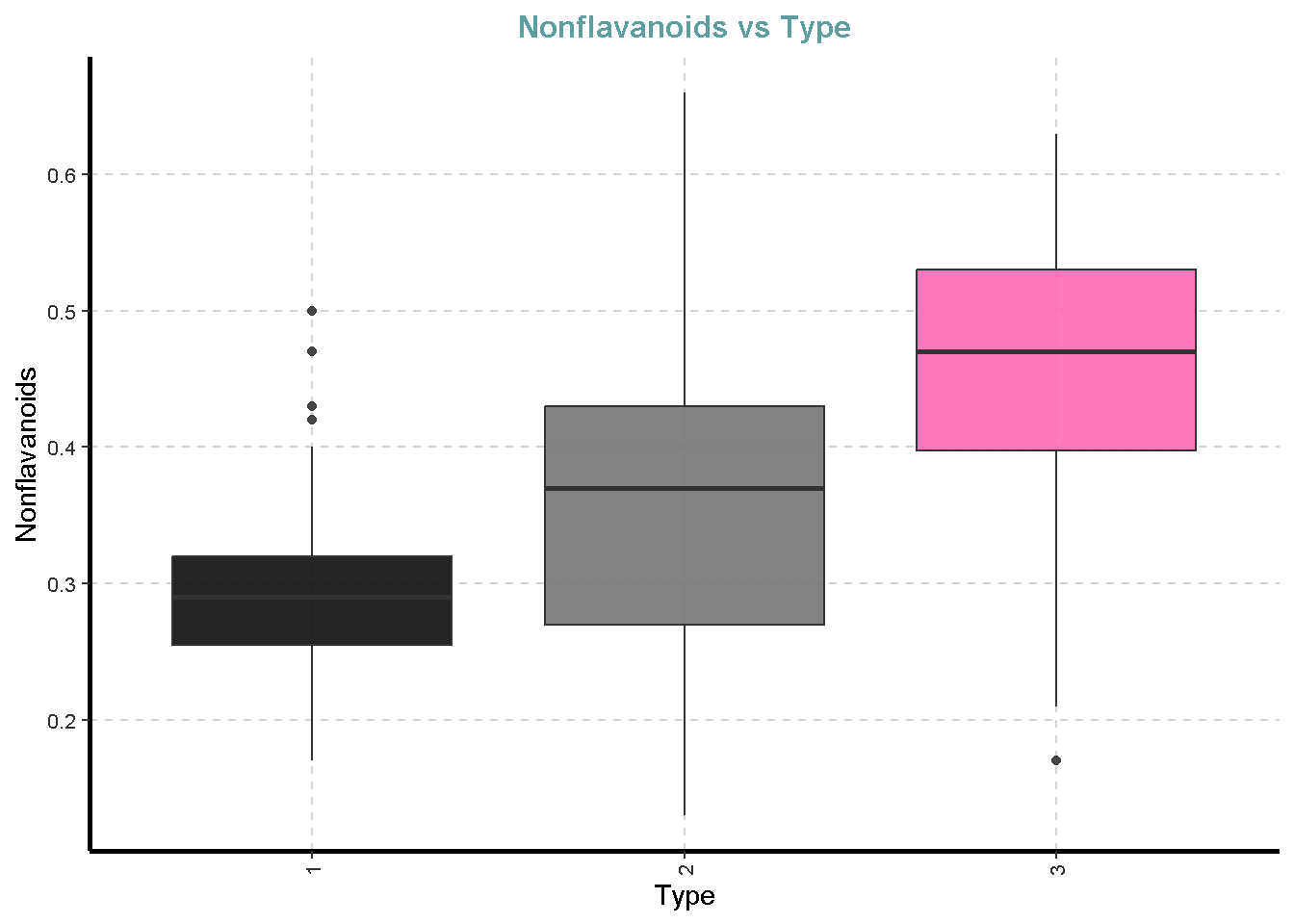
[[9]]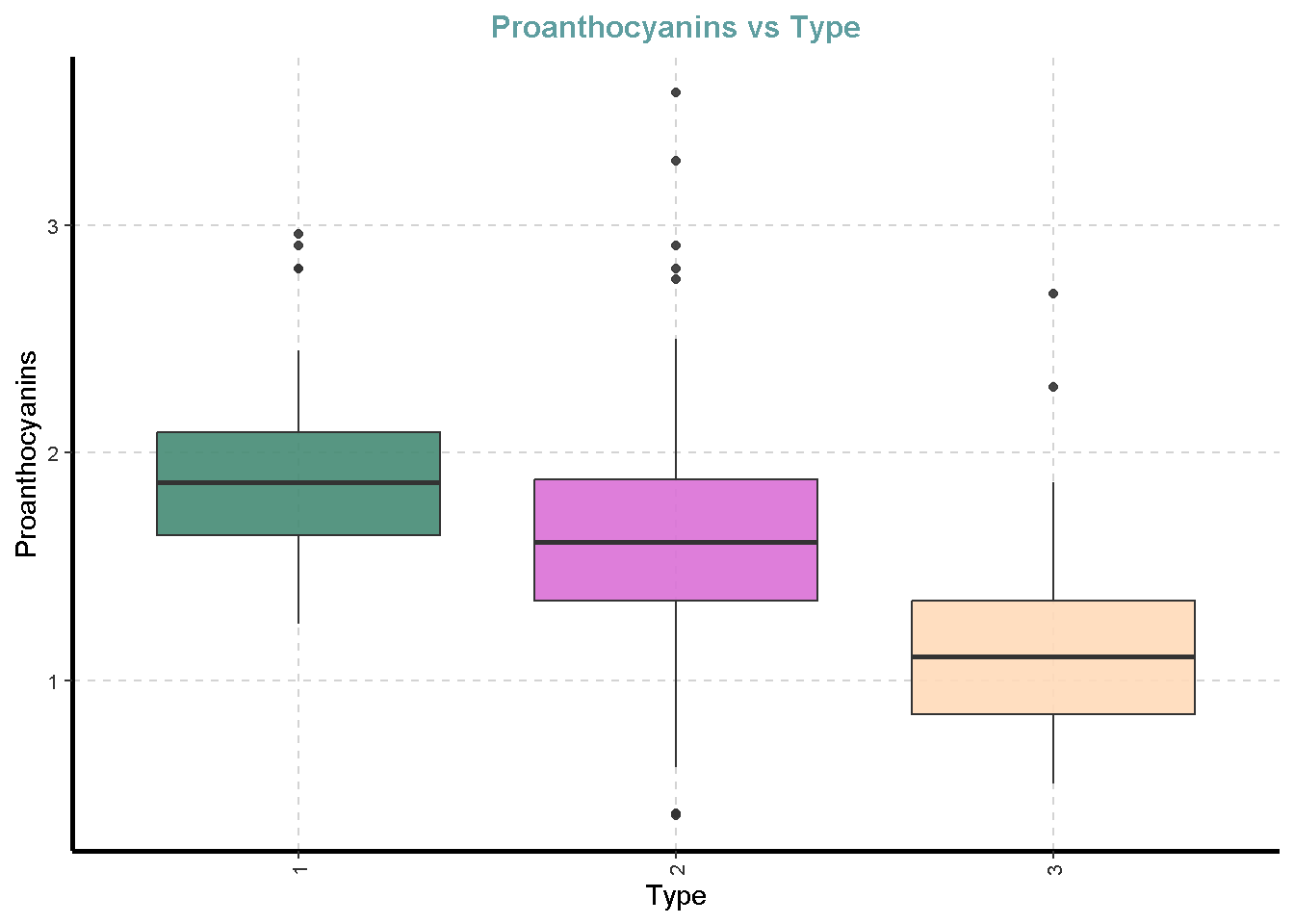
[[10]]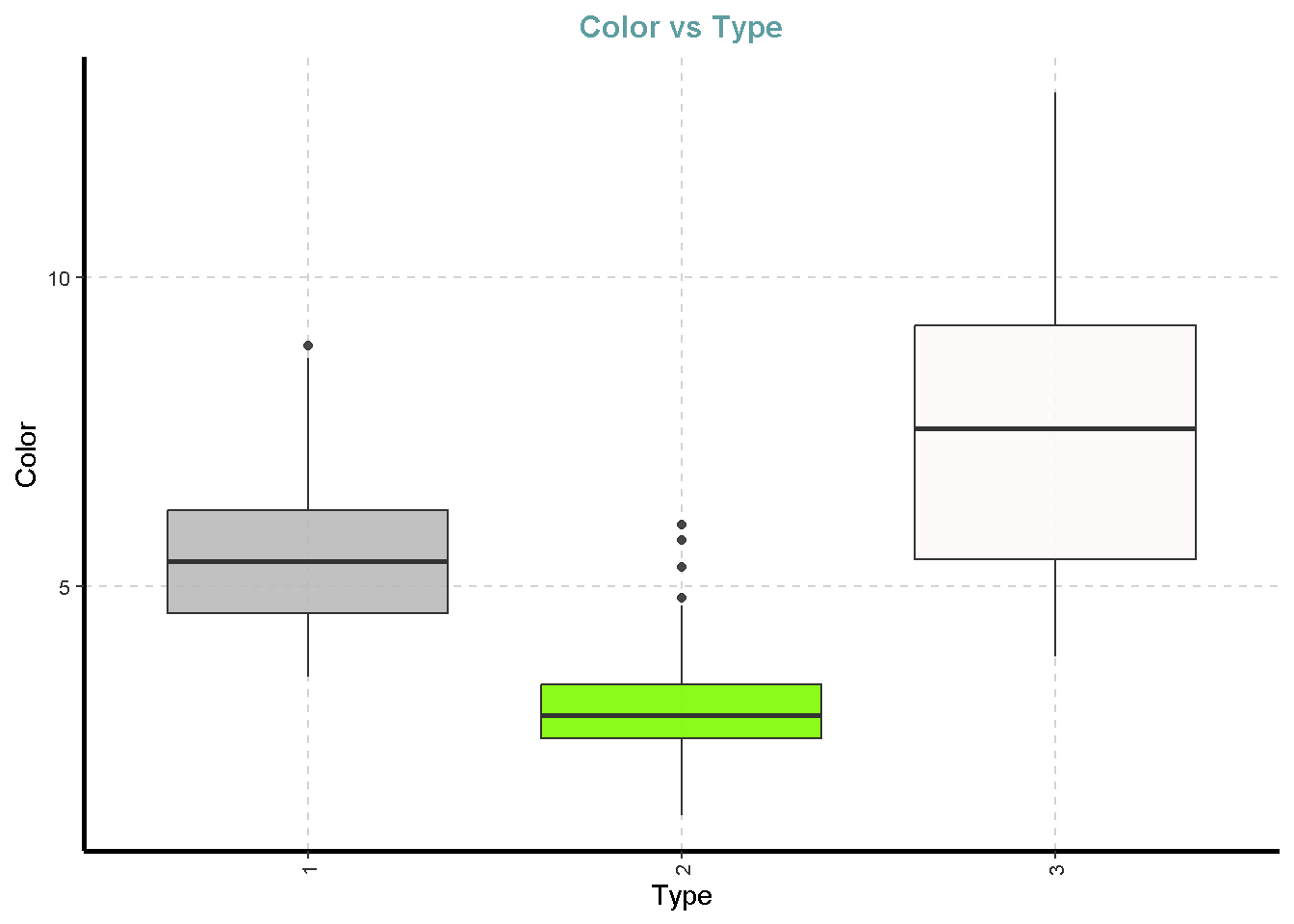
[[11]]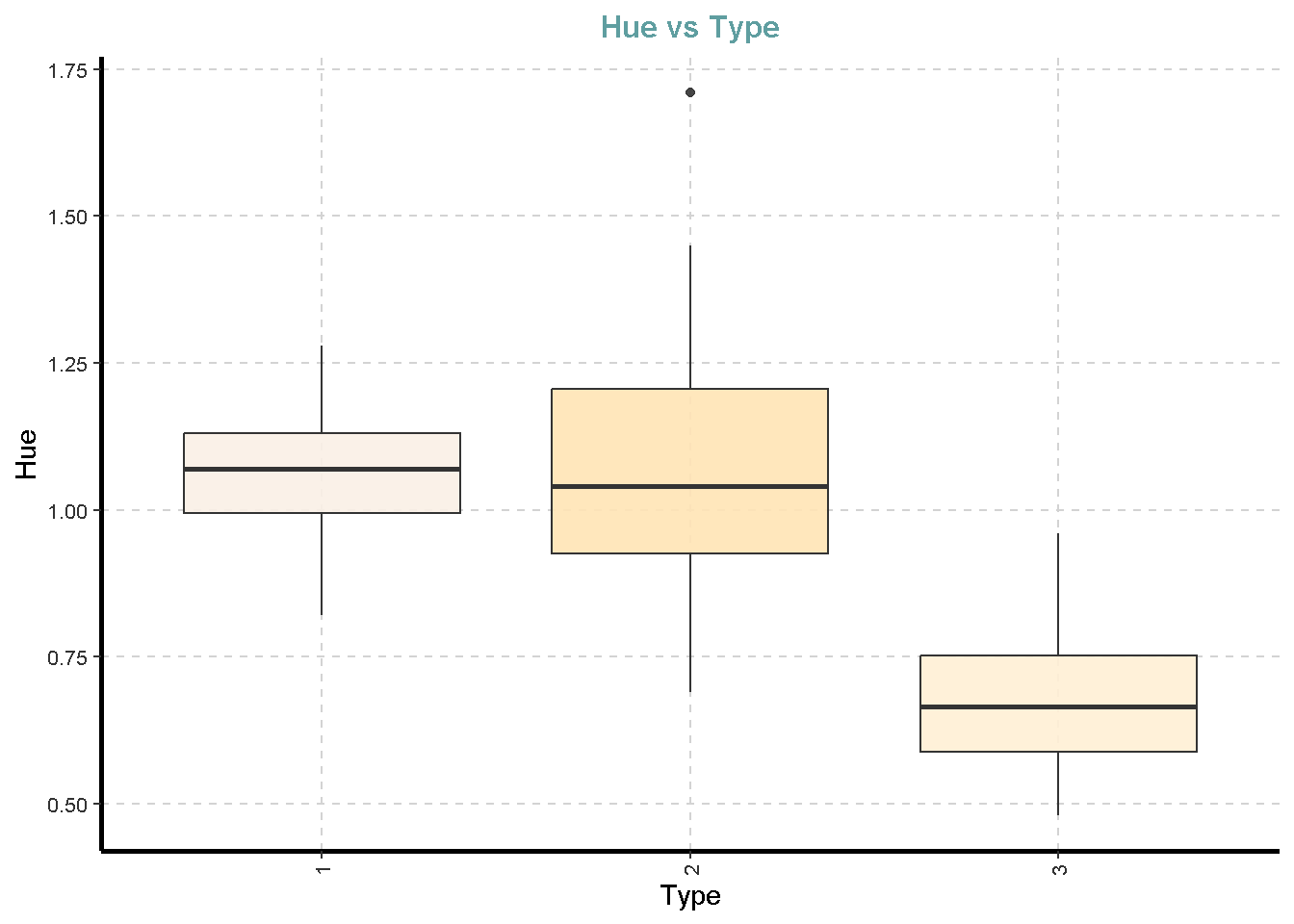
[[12]]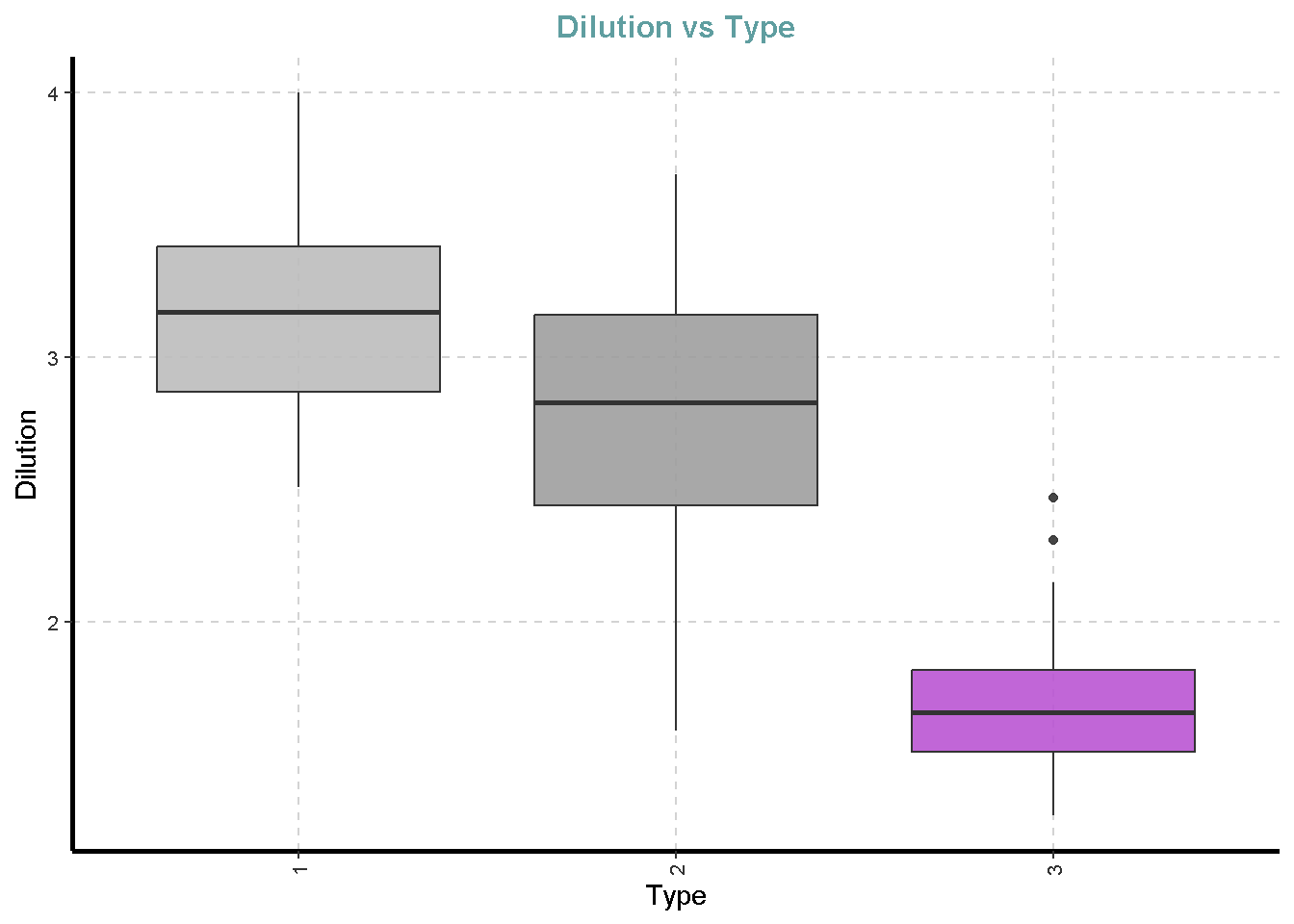
[[13]]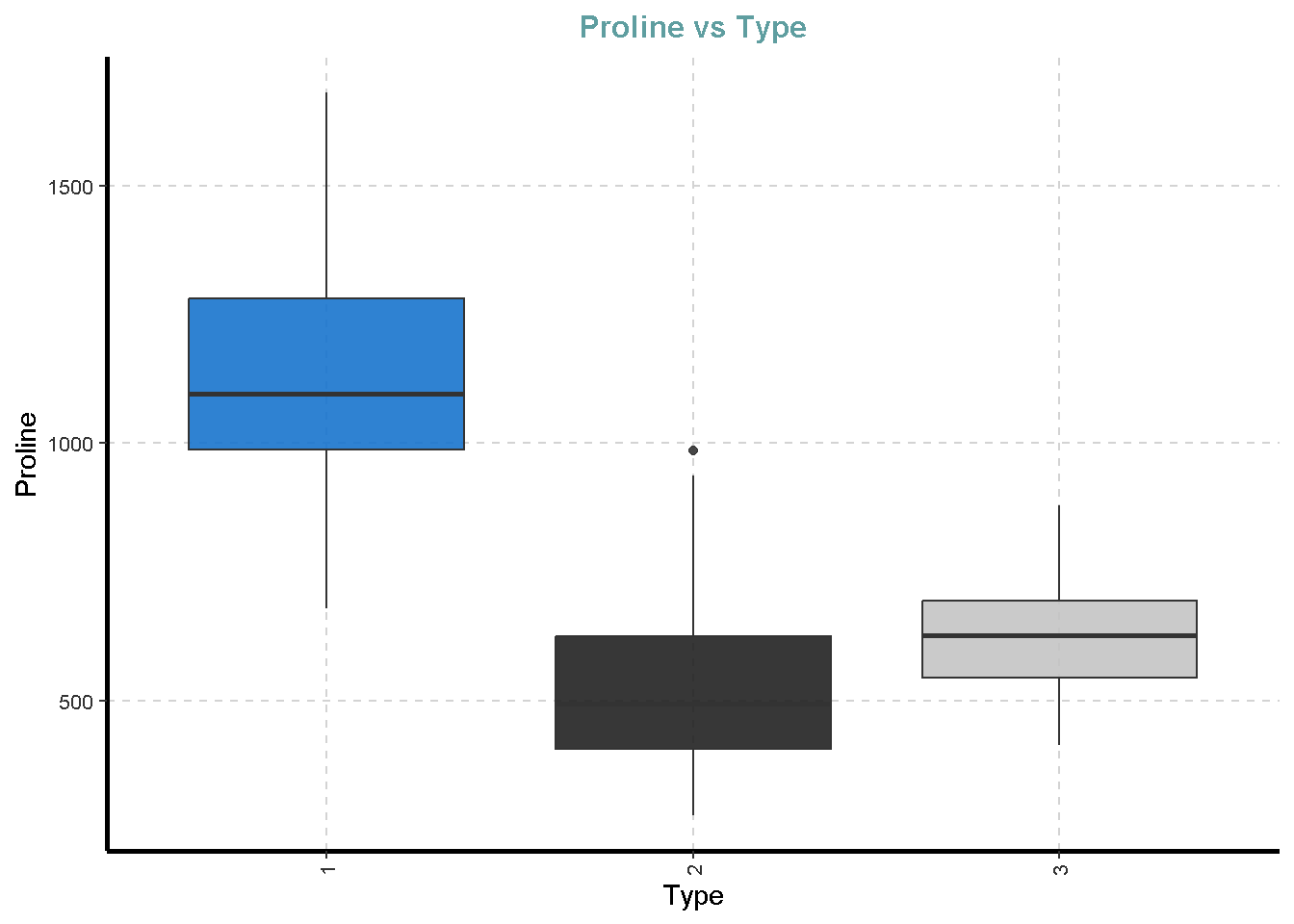
Deteksi Missing Value
df %>%
plot_missing(missing_only = FALSE,
ggtheme = theme_lares())Warning: `aes_string()` was deprecated in ggplot2 3.0.0.
ℹ Please use tidy evaluation idioms with `aes()`.
ℹ See also `vignette("ggplot2-in-packages")` for more information.
ℹ The deprecated feature was likely used in the DataExplorer package.
Please report the issue at
<https://github.com/boxuancui/DataExplorer/issues>.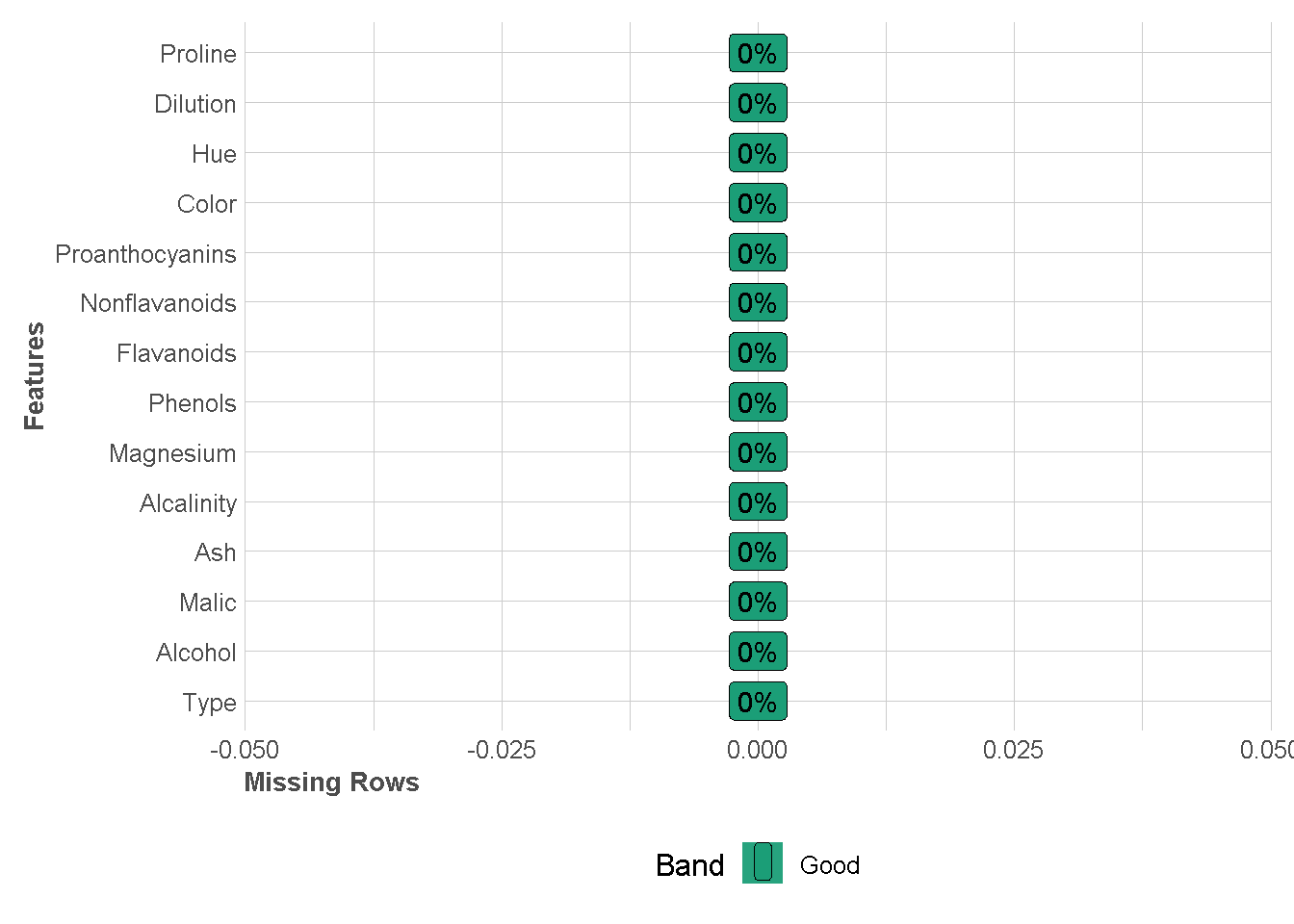
Analisis Diskirminant
Fungsi diskriminan dari Linear Discriminant Analysis (LDA) dapat dituliskan sebagai berikut:
\[ \boldsymbol{d}(\boldsymbol{x})=\log{P(\boldsymbol{y}=k|\boldsymbol{x})}=b_{0}+b_{1}\boldsymbol{x}_{1}+b_{2}\boldsymbol{x}_{2}+\ldots+b_{p}\boldsymbol{x}_{p} \]
dengan:
- \(\boldsymbol{y}\) adalah variabel respon dengan \(k\) kelas (kategori)
- \(\boldsymbol{d}(\boldsymbol{x})\) adalah skor diskriminan
- \(b_{0},b_{1},b_{2},\dots,b_{p}\) adalah koefisien atau bobot diskriminan
- \(\boldsymbol{x}_{1},\boldsymbol{x}_{2},\ldots,\boldsymbol{x}_{p}\) adalah variabel prediktor
Asumsi dari LDA antara lain adalah:
- Setiap amatan harus dikelompokkan hanya ke dalam satu kelas (kategori) saja
- Ragam dalam setiap kelas (kategori) adalah sama (equal variances)
- \(\boldsymbol{x}_{1},\boldsymbol{x}_{2},\ldots,\boldsymbol{x}_{p}\) berdistribusi normal ganda (multivariate normal distribution)
LDA dapat dilakukan bila terdapat perbedaan yang nyata antar kelas (kategori), sehingga pada tahap awal yang harus dilakukan adalah uji hipotesis bahwa tidak ada perbedaan kelas (kategori) di antara amatan.
Jika asumsi Ragam dalam setiap kelas (kategori) adalah sama tidak terpenuhi maka maka gunakan Quadratic Discriminant Analysis (QDA).
Note : Menurut Mattjik dan Sumertajaya pada buku Sidik Peubah Ganda, “umumnya sangat sulit sekali untuk dapat memenuhi persyaratan 2 dan 3, yang dalam praktek sering kali tidak diuji; hal mana akan membuat akurasi dari analisis dengan fungsi diskriminan akan berkurang. Namun demikian, fungsi diskriminan selalu menghasilkan estimasi yang kokoh (robust estimates) terutama yang berkaitan dengan prediksi kelas”.
Pemeriksaan Awal
Pada tahap ini kita akan memeriksa asumsi 2 dan 3 LDA serta memeriksa apakah ada perbedaan kelas (kategori) di antara amatan.
Uji Normal Ganda (Multivariate Normal) setiap kelas variabel respon
Hipotesis yang diuji adalah sebagai berikut
\[ \begin{aligned} H_0 &: \text{data menyebar normal ganda} \\ H_1 &: \text{data tidak menyebar normal ganda} \end{aligned} \]
Untuk menguji kenormalan ganda di R, bisa menggunakan fungsi mvn dari package MVN. fungsi mvn memiliki beberapa uji normal ganda yang bisa dilakukan. Pemilihan uji normal ganda bisa dilakukan melauli argumen mvnTest, seperti uji Mardia (mvnTest="mardia"), uji Henze-Zirkler(mvnTest="hz"), uji Royston (mvnTest="royston"),uji Doornik-Hansen (mvnTest="dh") dan uji energy mvnTest="energy". Argumen subset diisi dengan kolom data yang dijadikan sebagai variabel respon.
uji_normalGanda <- MVN::mvn(data = df,subset="Type",mvn_test = "hz")
uji_normalGanda$multivariate_normalityKarena nilai dari p-value dari $1 dan $3 adalah 0.198733 dan 0.3721148, yang mana lebih besar dari nilai \(\alpha= 0.05\) maka dapat disimpulkan bahwa tidak cukup bukti untuk menolak \(H_0\). Artinya untuk peubah-puebah prediktor pada wine tipe 1 dan tipe 3 berdistribusi normal ganda. Sementera itu, p-value dari $2 sangat kecil yaitu 4.160195e-06 yang mana lebih kecil dari nilai \(\alpha=0.05\). Artinya peubah-puebah penjelas pada wine tipe 2 tidak berdistribusi normal ganda.
Walaupun ada satu kelas yang tidak memenuhi asumsi normal ganda kita akan tetap lanjutkan menggunakan analisis diskriminan karena berdasarkan Matjik dan Sumertajaya fungsi diskriminan masih dapat menghasilkan kemampuan klasifikasi yang baik.
Uji Asumsi Kesamaan Ragam setiap kelas variabel respon
Hipotesis asumsi kesamaan ragam
\[ \begin{aligned} H_0 &: \text{ragam antar populasi sama} \\ H_1 &: \text{ragam antar populasi tidak sama} \end{aligned} \]
Uji kesamaan ragam bisa dilakukan dengan menggunkan fungsi boxM yang berasal dari package heplots. Fungsi ini hanya membutuhkan 2 argumen, yaitu data dalam bentuk data.frame atau matrix tanpa kolom gerombol (dalam hal ini kolom Type) dan vektor gerombol yang diperoleh dari data (dalam hal ini kolom Type).
uji_ragam <- heplots::boxM(Y=df %>% dplyr::select(-Type),
group=df$Type)
tidy(uji_ragam)Karena nilai dari p-value dari uji Box’s M adalah kurang dari 2.891851e-59, yang mana lebih kecil dari nilai \(\alpha\) 0.05 maka dapat disimpulkan bahwa cukup bukti untuk menolak \(H_0\). Artinya ragam setiap kelas variabel respon tidak sama. Hal ini berarti model yang lebih cocok digunakan adalah QDA.
Namun pada tutorial ini kita akan mencoba menggunakan keduanya (LDA dan QDA) dan akan kita bandingkan menggunakan metric yang lain.
Menyiapkan Pembagian Data
K-fold Cross Validation
K-fold Cross Validation adalah prosedur pengambilan sampel berulang yang melibatkan pembagian dataset menjadi K bagian yang sama besar atau hampir sama besar, yang disebut fold (lipatan). Ide dari metode ini adalah untuk training dan testing model klasifikasi sebanyak K kali, menggunakan fold(lipatan) yang berbeda sebagai testing data dalam setiap ulangan sambil menggunakan lipatan K-1 yang lain sebagai training data.
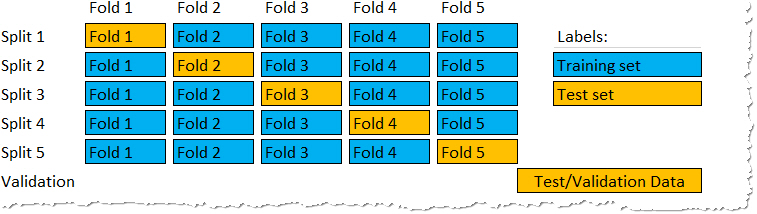
Pemilihan nilai K tergantung pada ukuran dataset dan trade-off antara waktu komputasi dan bias validasi. Nilai umum untuk K adalah 5 dan 10, tetapi kita dapat bereksperimen dengan nilai yang berbeda untuk menemukan keseimbangan terbaik untuk masalah spesifik.
set.seed(345)
folds <- vfold_cv(df, v = 10,strata = "Type" )Mendefinisikan Model
Linear Discriminant
disc_linear <- discrim_linear()%>%
set_engine(engine = "MASS") %>%
set_mode("classification")Quadratic Discriminant
disc_quad <- discrim_quad() %>%
set_engine(engine = "MASS") %>%
set_mode("classification")Mendefinisikan praproses data
Pendefinisikan praproses data dapat dilakukan dengan fungsi recipe.
basic_recipe <- recipe(Type~.,data =df)
basic_recipe ── Recipe ──────────────────────────────────────────────────────────────────────── Inputs Number of variables by roleoutcome: 1
predictor: 13Menggabungkan tahap praproses data dan model
models <- list(disc_linear=disc_linear,
disc_quad =disc_quad)
preproc <- list(basic_recipe=basic_recipe)
wf_set <- workflow_set(preproc = preproc,models = models,
cross = TRUE)Training Models
train_models <- workflow_map(wf_set,
fn="fit_resamples",
resamples = folds,
control=control_resamples(save_workflow = TRUE),
metrics=metric_set(sensitivity,
specificity,
bal_accuracy,
f_meas),
seed = 2123)Evaluasi Model
autoplot(train_models,type="wflow_id",
rank_metric = "bal_accuracy")+
scale_y_continuous(limits = c(0,1)) +
theme_lares() +
# memindahkan legend ke bawah
theme(legend.position = "bottom")+
# legend menjadi 2 baris
guides(color = guide_legend(nrow = 2))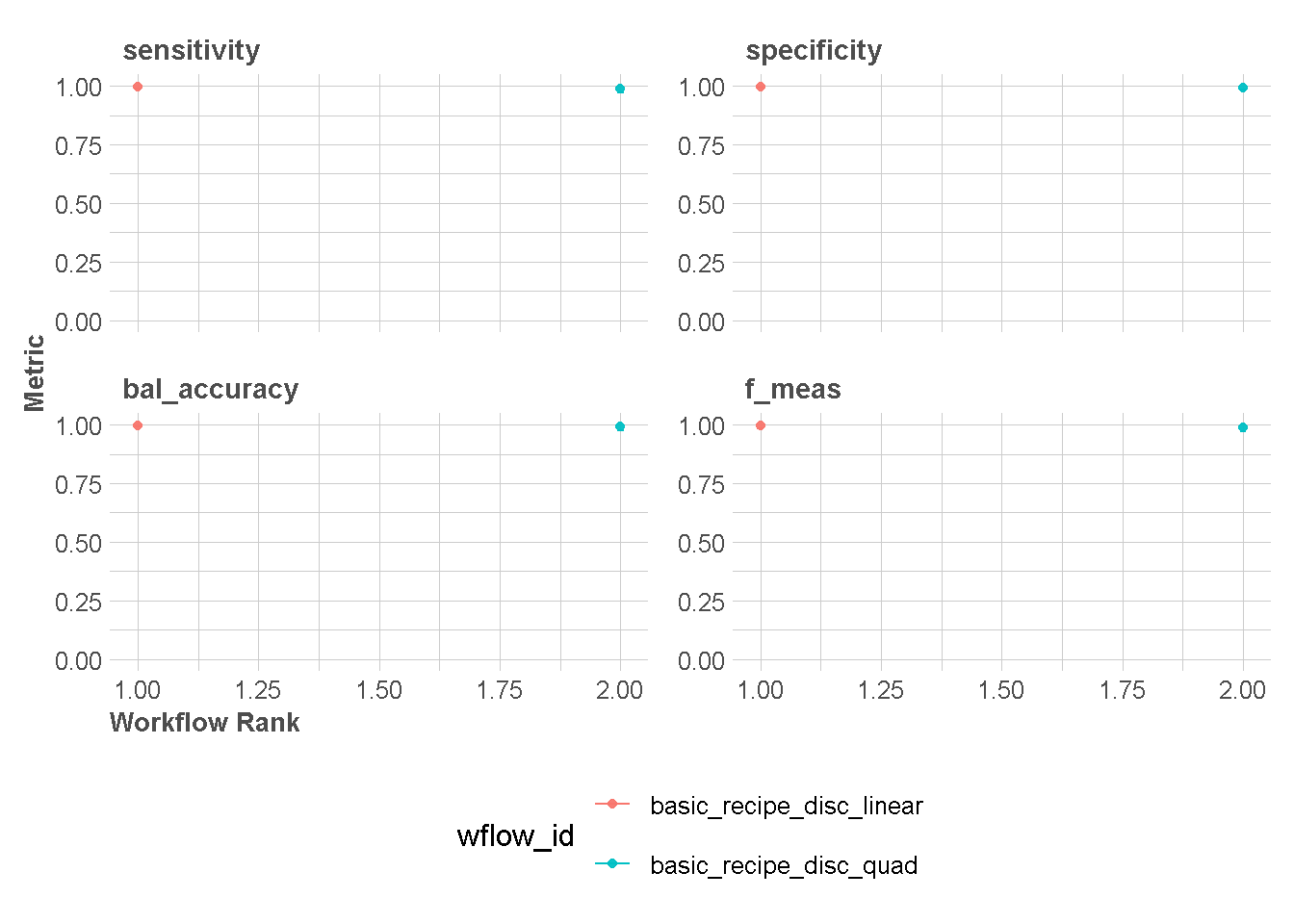
rank_results(train_models,rank_metric = "bal_accuracy") %>%
filter(.metric=="bal_accuracy") %>%
dplyr::select(wflow_id,.metric,mean,std_err,rank)rank_results(train_models,rank_metric = "f_meas") %>%
filter(.metric=="f_meas") %>%
dplyr::select(wflow_id,.metric,mean,std_err,rank)Menentukan Model terbaik dari Training
mod_best1 <- fit_best(x = train_models,metric = "bal_accuracy")
mod_best1══ Workflow [trained] ══════════════════════════════════════════════════════════
Preprocessor: Recipe
Model: discrim_linear()
── Preprocessor ────────────────────────────────────────────────────────────────
0 Recipe Steps
── Model ───────────────────────────────────────────────────────────────────────
Call:
lda(..y ~ ., data = data)
Prior probabilities of groups:
1 2 3
0.3314607 0.3988764 0.2696629
Group means:
Alcohol Malic Ash Alcalinity Magnesium Phenols Flavanoids
1 13.74475 2.010678 2.455593 17.03729 106.3390 2.840169 2.9823729
2 12.27873 1.932676 2.244789 20.23803 94.5493 2.258873 2.0808451
3 13.15375 3.333750 2.437083 21.41667 99.3125 1.678750 0.7814583
Nonflavanoids Proanthocyanins Color Hue Dilution Proline
1 0.290000 1.899322 5.528305 1.0620339 3.157797 1115.7119
2 0.363662 1.630282 3.086620 1.0562817 2.785352 519.5070
3 0.447500 1.153542 7.396250 0.6827083 1.683542 629.8958
Coefficients of linear discriminants:
LD1 LD2
Alcohol -0.403399781 0.8717930699
Malic 0.165254596 0.3053797325
Ash -0.369075256 2.3458497486
Alcalinity 0.154797889 -0.1463807654
Magnesium -0.002163496 -0.0004627565
Phenols 0.618052068 -0.0322128171
Flavanoids -1.661191235 -0.4919980543
Nonflavanoids -1.495818440 -1.6309537953
Proanthocyanins 0.134092628 -0.3070875776
Color 0.355055710 0.2532306865
Hue -0.818036073 -1.5156344987
Dilution -1.157559376 0.0511839665
Proline -0.002691206 0.0028529846
Proportion of trace:
LD1 LD2
0.6875 0.3125 Interpretasi Model Terbaik
coefficient <- mod_best1 %>%
extract_fit_engine() %>%
coef() %>%
as.data.frame() %>%
rownames_to_column(var = "Predictors")
coefficient## custom function
lda_equation <-function(x,names){
str_c(str_c("(",x,")",
names,collapse = "+"
)
)
}coefficient %>%
mutate(across(where(is.numeric),function(x) round(x,digits = 2))) %>%
summarise(D1=lda_equation(LD1,Predictors),
D2=lda_equation(LD2,Predictors)) %>%
pivot_longer(c(D1,D2),names_to = "discriminant",
values_to="equation"
)## custom function
autoplot.lda<-function(object,response,data){
object %>%
predict(newdata = data) %>%
magrittr::extract2("x") %>%
as_tibble() %>%
bind_cols(data %>% dplyr::select(all_of(response))) %>%
ggscatter(x="LD1",y="LD2",color = response)
}mod_best1 %>%
extract_fit_engine() %>%
autoplot(response="Type",data=df)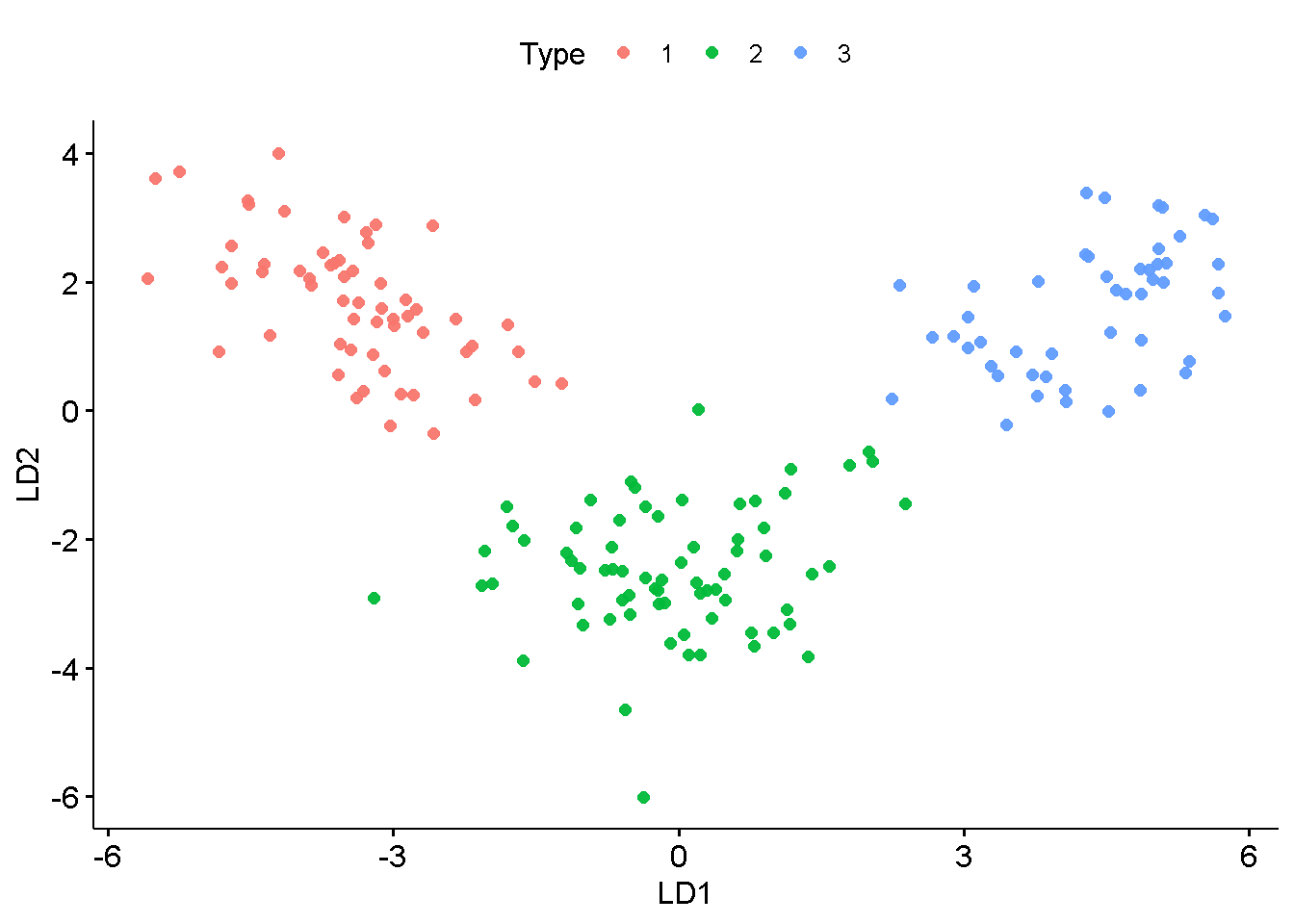
Prediksi Data baru
Berikut kita generate data baru dummy
set.seed(1234)
data_baru <- df %>%
slice_sample(n = 2,by = Type) %>%
dplyr::select(-Type)
data_barupred_data_baru1 <- mod_best1%>%
predict(new_data = data_baru)pred_data_baru1